Идентификация генов – основная задача нового этапа развития генетики
Контрольная работа
Идентификация генов - основная задача
нового этапа развития генетики
Содержание
1. Эволюция представлений о гене и понятия идентификации гена
. Основные методы идентификации генов растений
. Позиционное клонирование (выделение) генов, маркированных
мутациями
. Идентификация генов, маркированных точковыми мутациями,
методом TILLING
. Выделение генов, маркированных делециями методом геномного
вычитания
. Выделение генов, маркированных делециями, с помощью метода
Delet-a-gen
Литература
1. Эволюция представлений о гене и понятия идентификации гена
Проблема гена являлась центральной на всем протяжении существования
генетики как экспериментальной науки, и представление о гене постепенно
усложнялось и эволюционировало. До открытия в 1944 году в результате работ
Эйвери и Мак-Леода по трансформации у бактерий роли ДНК как материального
носителя наследственности и определения ее структуры основным нерешенным
вопросом было физико-химическое наполнение понятия гена. Начиная с 50-х годов
прошлого столетия, основные акценты в изучении гена сместились в сторону
изучения структурно-функциональной организации гена. Начало нового тысячелетия
ознаменовалось серьезными успехами в определении полной нуклеотидной
последовательности генома целого ряда видов про- и эукариот, включая человека,
дрозофилу, мышь, высшее растение Arabidopsis thaliana (www.arabidopsis.org). Сегодня завершена работа по секвенированию генома
двух подвидов риса Oriza sativa (#"868572.files/image001.gif">
Рисунок 1. Метод позиционного клонирования генов (объяснение в тексте)
На этом этапе (рис.1б) осуществляют поиск фрагментов ДНК, перекрывающих
район хромосомы от маркера ПДРФ1 до ПДРФ2, с использованием двух библиотек ДНК
с разными сайтами клонирования. В одной из них находят клон, гибридизующийся с
ПДРФ1-маркером2, а затем этот клон служит ДНК-зондом для выявления перекрывающегося
клона другой библиотеки. Зондом для каждого следующего клона служит короткий
концевой Р32-фрагмент ДНК предыдущего идентифицированного клона3. Из этого
нового клона получают затем ДНК-зонд, используемый для нахождения другого
перекрывающегося клона первой библиотеки, и так до тех пор, пока не будет
найден клoн, имеющий в своем составе маркер
ПДРФ2. Очевидно, что в составе выявленных клонов обязательно будет содержаться
и искомый ген.
На заключительном этапе осуществляют поиск последовательности искомого
гена в выявленных клонах (клоне). С этой целью фрагменты ДНК из клонов (это,
как правило, крупные от 100 до 800 000 тпн фрагменты, клонированные в BAC или YAC векторах), подвергают рестрикции. Полученные
фрагменты включают в состав вектора, содержащего Т-ДНК область от Ti-плазмиды A. tumefaciens, и
клонируют в клетках E.coli. После перенесения размноженных
векторных плазмид в клетки A.tumefaciens (с помощью электропорации или других
методов), осуществляют трансформацию растений (рис. 1в).
О наличии в составе фрагмента ДНК искомого гена судят по его способности
функционально комплементировать мутацию, т.е. восстанавливать у мутантных
растений фенотип дикого типа. Используя компьютерные базы данных, анализируют
функции открытых рамок считывания, расположенных в найденном клоне ДНК.
Сопоставляя полученную информацию с фенотипическими особенностями мутантов,
определяют рамку считывания, соответствующую искомому гену. Дополнительным
подтверждением выделения искомого гена может служить анализ нуклеотидной последовательности
у нескольких мутантов по данному гену. У всех мутантов должны обнаруживаться
определенные изменения последовательности нуклеотидов в исследуемом гене,
способные вызвать нарушение его функции.
. Идентификация генов, маркированных точковыми мутациями, методом TILLING
Точковые мутации открывают возможности и для изучения генов методами
"обратной" генетики. Главное достоинство точковых мутаций - их
высокая частота. Так, например для A.thaliana подсчитано, что для нахождения с
вероятностью 99% мутации в гене размером 1 т.п.н. требуется: на основе
инсерционого мутагенеза - 550 000 линий, на основе делеционных мутантов - 130
397 линий, а на основе химического мутагенеза с использованием ЭМС - всего 4
000 -10 000 индивидуальных семей М2 поколения).
Суть метода TILLING
заключается в получении мутаций в интересующем исследователя гене с известной
нуклеотидной последовательностью. На первом этапе после обработки мутагеном
семян (у растений чаще всего используется этилметансульфонат, действие которого
на ДНК высоко специфично и заключается в индукции транзиций ГЦАТ)
получают популяцию растений М2, из которых выделяют ДНК и проводят
индивидуальный сбор семян М3. После объединения в небольшие пулы (ДНК с 3-8
растений М2), ДНК используется для проведения ПЦР со специфическими праймерами
(рис. 2).
На втором этапе среди продуктов ПЦР, полученных от разных пулов растений
М2, осуществляется поиск тех, которые несут точковые мутации. Выявление мутаций
основано на формировании гетеродуплексов у гетерозигот по мутации и
осуществляется с помощью одного из двух способов (рис. 2):
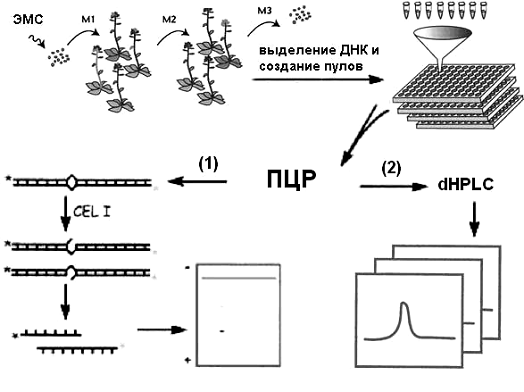
Рисунок 2. Схема получения семян М2 и создания пулов ДНК для работы
методом TILLING
(2) Мутации, вызывающие неправильное спаривание оснований (mismatch), можно выявить и с помощью
эндонуклеазы сельдерея CEL1,
которая распознает неправильно спаренные основания всех типов. Рестрикции
подвергают амплифицированные фрагменты ДНК. Для амплификации используют пары
специфических праймеров, причем праймеры в прямой ориентации включают
флуорецирующую метку 6 - FAM
(6-carboxyfluorescein), придающую синюю окраску нити ДНК,
а праймеры в обратной ориентации включают другую метку - ТЕТ (4,7,2,7-tetrachloro-6-carboxyfluorescein), окращивающую нить ДНК в зеленый
цвет. Амплифицированные продукты инкубируют с эндонуклеазой CEL1, которая разрезает гетеродуплексную
ДНК в местах неправильно спаренных оснований с 3′-конца при pH от 6 до 9.
Продукты рестрикции можно выявить с помощью денатурирующего электрофореза
в акриламидном геле с высоким разрешением. Различия в окраске концов двойных
нитей ДНК позволяют легко визуально отличить комплементарные нити от
амплифицированных артефактов (рис. 3).
Поскольку размер одного пула может составлять смесь ДНК от 8 растений М2,
то при наличии большой электрофоретической камеры, например с 96 дорожками,
можно одномоментно анализировать на наличие мутации популяцию М2, состоящую из
768 растений. После выявления продуктов ПЦР, содержащих гетеродуплексы, можно
найти исходные растения М2, содержащие мутации, и в их семенном потомстве
провести анализ фенотипических изменений, необходимый для установлении функции
гена на уровне организма.
В настоящее время для модельных растительных объектов (арабидопсиса,
люцерны, риса) созданы специальные научные консорциумы, которые осуществляют
широкомасштабные эксперименты по химическому мутагенезу, направленные на
получение индуцированных мутаций по всем генам этих видов. В рамках этих
проектов предоставляются и платные услуги по выявлению индуцированных
этилметансульфонатом мутаций в любом интересующем исследователя гене этих видов
растений. После оформления заказа, консорциум проводит анализ 3000 растений М2
на наличие мутаций в данном районе гена и предоставляет заказчику семена растений,
содержащих мутацию в интересующем гене. Исследование фенотипа мутантов
позволяет исследовать функцию гена на уровне целого растения, т.е. завершить
выполнение задачи по идентификации гена.
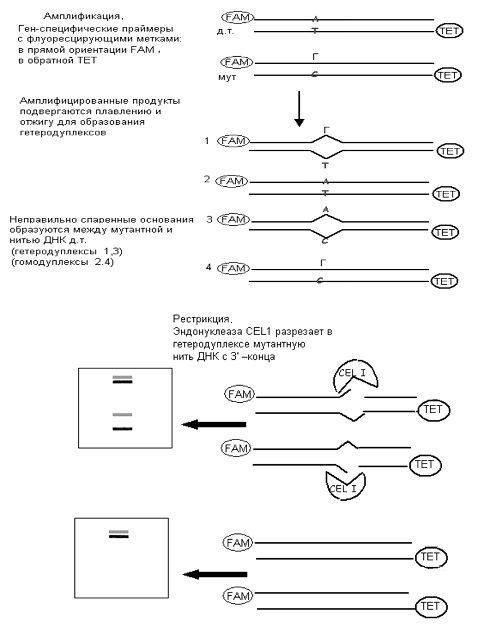
Рисунок 3. Выявление мутаций с помощью эндонуклеазы CEL1. Показаны основные этапы метода TILLING: амплификация пула ДНК со
специфическими праймерами к исследуемому гену, рестрикция продуктов ПЦР
эндонуклеазой CEL1 и анализ продуктов рестрикции с
помощью электрофореза геле (из #"868572.files/image004.gif">
Рисунок 4. Схема выделения генов методом геномного вычитания. В ДНК
мутанта делетирован район «В», поэтому фрагмент ДНК из растений дикого типа,
соответствующий району «В», не способен гибридизоваться с меченой биотином ДНК
мутанта. После пропускания гибридизационной смеси через колонку с покрытыми
авидином гранулами в растворе остаются лишь фрагменты ДНК из растений дикого
типа, соответствующие району делеции (фрагменты В).
Последовательные этапы метода геномного вычитания изображены на рис. 4.
Геномную ДНК мутанта фрагментируют ультразвуковой обработкой и после меченья
биотином гибридизуют с избытком обработанной рестрикционной эндонуклеазой ДНК
из растений дикого типа. Фрагменты, которые не прогибридизовались с ДНК
мутанта, соответствуют району делеции. Эти фрагменты не содержат биотиновой
метки, поэтому их выделяют, пропустив смесь через колонку с гранулами,
покрытыми авидином. Авидин, образуя прочные комплексы с биотином, связывает все
меченые фрагменты, в том числе и гибридные. В растворе остаются только не
меченые фрагменты ДНК, соответствующие району делеции. После их выделения и
амплификации, их клонируют и используют в качестве проб для поиска
полноразмерной копии гена в библиотеках ДНК из растений дикого типа.
Этап клонирования не является обязательным, поскольку в качестве пробы
для скрининга библиотек можно использовать и меченые продукты ПЦР. Для видов с
полностью или частично секвенированными геномами вместо скрининга библиотек для
обнаружения полноразмерных копий гена можно использовать компьютерный анализ.
Для его проведения достаточно определить последовательность нуклеотидов амплифицированного
фрагмента растительной ДНК и с помощью программ BLASTA или FASTA
осуществить поиск в секвенированном геноме растения гомологичных
последовательностей ДНК, а, следовательно, и исследуемого гена. Исследование
структуры гена позволяет выяснить молекулярную функцию гена и, таким образом,
завершить идентификацию гена, маркированного делецией.
. Выделение генов, маркированных делециями, с помощью метода Delet-a-gen
Метод Delet-a-gen, так же как
метод TILLING, относится к методам «обратной» генетики, поскольку направлен на
поиск делеций в генах с известной нуклеотидной последовательностью. Семена
растений дикого типа облучают быстрыми нейтронами, проращивают и получают
растения М1, с которых индивидуально собирают семена М2. Из 10 проростков индивидуальной
семьи М2 выделяют ДНК, после чего берутся аликвоты из разных семей и
составляются пулы ДНК. Самый малочисленный пул составляется из аликвот ДНК от
18 семей, второй пул состоит из 36 семей, суперпул из 288, и мегапул из 2592
семей. Пулы ДНК используют для проведения ПЦР с парой специфических праймеров
по фланкирующим последовательностям целевого гена. О присутствии делеции в гене
судят по появлению продуктов ПЦР меньшего размера, чем у дикого типа. С помощью
подбора условий ПЦР (сокращение времени элонгации) добиваются преимущественной
амплификации делетированной (укороченной) копии гена, что позволяет выявлять
делеции среди мегапулов ДНК, в которых содержание ДНК от растений, не несущих
делецию, в тысячу раз выше, чем от делеционных мутантов.
После выявления делеции в мегапуле, проводят анализ суперпулов и пулов,
что в конечном счете позволяет найти исходные растения М2 с делецией,
исследовать фенотипы потомков и функцию гена на уровне растения.
ген клонирование мутация
Литература
1. Гапеева
Т.А., Огаркова О.А., Тарасов В.А., Волотовский И.Д. 2005. Новые вектора для
трансформации двудольных растений // Генетика. 31, 8: 1085 - 1091.
2. Дрейпер
Дж., Скотт Р., Армитидж Ф., и др. 2011. Генная инженерия растений: Лабораторное
руководство // М.: Мир. 408 c.
3. Лутова
Л.А., Павлова З.Б., Иванова М.М. 2008. Агробактериальная трансформация как
способ изменения гормонального метаболизма у высших растений // Генетика. 34,
2: 165-182.
4. Маниатис
Т., Фрич Э., Сэмбрук Дж. 1984. Молекулярное клонирование // М.: Мир. 479 с.
6. Пирузян
Э.С. 1988. Основы генетической инженерии растений // М.: Наука. 303 с.
7. Рыбчин
В.Н. 2009. Основы генетической инженерии // С.-П.: СПбГТУ. С. 1081-1087.
. Филипенко
Е.А., Филипенко М.Л., Мурашева С.В., Дейнеко Е.В., Загорская А.А., Сидорчук
Ю.В. 2000. Анализ сайтов встраивания Т-ДНК у трансгенных растений табака с
мутантным фенотипом // Докл. Акад. Наук. 370: 273-276.
9. Чумаков
М.И. 2011. Механизм агробактериальной трансформации // Саратов: Слово. 256 с.
10. An
G., Watson B.D., Chiang C.C. 1986. Transformation of tobacco, tomato, potato
and Arabidopsis thaliana using a binary Ti-vector system // Plant Physiol. 81,
1: 301-305.
. Aufrsatz
W., Mette M.F., van der Winder J., Matzke A., Matske M. 2002. RNA-directed DNA methylation in
Arabidopsis // Proc. Natl Acad Sci USA. 99: 16, 499-16,506.
. Azpiroz-Leehan
R., Feldmann K.A. 1997. T-DNA mutagenesis in Arabidopsis: going back and forth
// Trends in Genet. 13, 4: 152-156.
. Bartel
D.P. 2004. MicroRNAs: genomics, biogenesis, mechanism, fnd function // Cell.
116. 281-297.
. Bechtold
N., Ellis J., Pelletier G. 2003. In planta Agrobacterium- mediated gene
transfer by infiltration of adult Arabidopsis thaliana plants // Life sciences.
316: 1194 - 1199.
. Beclin
C., Boutet S., Waterhouse P., Vaucheret H. 2012. A branched pathway for
transegene-induced RNA silencing in plants // Curr. Biol. 12: 684-688.
. Bernstein
E., Caudy A.A., Hammond S.M., Hannon G.J. 2001.Pole for a bidentate
ribonuclease in the initiation step of RNA interference // Nature. 409: 363-366.
. Birchler
J.A., Pal Bhadra M., Bhadra U. 2010. Making noise about silence : repression of
repeated genes in animals. // Curr Opin Genet Dev. 10: 211-216.
. Blackwood
E.M., Kadonaga J.T. 2008. Going the distance: a current view of enhancer action
// Science. 281: 61-63.
19. Bouchez
D., Hofte H. 1998. Functional genomics in plants // Plant Physiol. 118:
725-732.
20. Brunaud V., Balzergue
S., Dubreucq B., Aubourg S., Samson F., Chauvin S., Bechtold N., Cruaud C.,
DeRose R., Pelletier G., Lepiniec L., Caboche M., Lecharny A. 2002. T-DNA
integration into the Arabidopsis genome depends on sequences of pre-insertion
sites // EMBO Reports. 3, 12: 1152-1157.