Письменный перевод с английского языка на русский 'DNA Replication in Archaea, the Third Domain of Life'
МИНИСТЕРСТВО ОБРАЗОВАНИЯ И НАУКИ
РОССИЙСКОЙ ФЕДЕРАЦИИ
Федеральное государственное бюджетное
образовательное учреждение высшего профессионального образования
«Кубанский государственный
университет»
Дополнительная профессиональная
образовательная программа профессиональной переподготовки для получения
дополнительной квалификации
«Переводчик в сфере профессиональной
коммуникации»
ОТЧЕТ ПО ПЕРЕВОДЧЕСКОЙ ПРАКТИКЕ
Письменный перевод с английского
языка на русский
«DNA Replication in
Archaea, the Third Domain of Life»
Выполнил: Юхновский С.А.
Проверила: Спасова М.В.
Краснодар, 2013
TABLE OF CONTENTS
Essay
English text
1. Introduction
2. Replication origin
3. How does Cdc6/Orc1 recognize oriC?
. MCM helicase
. Recruitment of Mcm to the oriC region
. GINS
. Primase
. Single-stranded DNA binding protein
. DNA polymerase
. PCNA and RFC
. DNA ligase
. Flap endonuclease 1 (FEN1)
. Summary and perspectives
I. Russian Translation
II. Linguistic analysis of the Text
Bibliography
СОДЕРЖАНИЕ
Эссе
Английский
текст
. Введение
2. Инициация
репликации
. Как
распознают Cdc6/Orc1 в ORIC?
4. MCM
геликаза
5. Комплектование
MCM в области ORIC
. GINS
7. Праймазы
. Одноцепочечный
ДНК-связывающий белок
. ДНК-полимераза
10.
PCNA и RFC
11.
ДНК-лигазы
12.
Флэп-эндонуклеаза 1
(FEN1)
13.
Результаты и перспективы
III. Русский
перевод
IV. Лингвистический
анализ текста
Библиография
Глоссарий
Essay
My graduation paper includes thirteen parts, which are
excerpts of two books. The first part is "DNA replication in Archaea, the
Third Domain of Life", which gives a simple explanation of the molecular
mechanism of DNA replication for solving biological problems. The source for
this part was the book "Mechanisms of DNA replication", written and
edited by Stewart. The second part is the "Replication origin", which
describes the initiation factor, now referred to as a replication origin. The
third part is the "How does Cdc6/Orc1 recognize oriC", which
describes understanding how the Cdc6/Orc1 protein recognizes the oriC region.
The fourth part is the "MCM helicase", which gives an understanding
of the functions of the MCM helicase. The fifth part is the " Recruitment
of Mcm to the oriC region", which solved an another important question -
is how MCM is recruited onto the unwound region of oriC. The sixth part is the
"GINS", which describes the main function of the GINS complex. The
seventh part is the "Primase", which describes the short
oligonucleotide, that required for the synthesis as a primer. The eighth part
is the "Single-stranded DNA binding protein", which describes an
important factor to protect the unwound single-stranded DNA from nuclease
attack, chemical modification, and other disruptions during the DNA replication
and repair processes. The ninth part is the "DNA polymerase", which
describes a fundamental ability of DNA polymerases. The tenth part is the
"PCNA and RFC", which provides an understanding of the functions of
these protein structures. The eleventh part is the "DNA ligase",
which describes how this enzyme to catalyze phosphodiester bond formation via
three nucleotidyl transfer steps. The twelfth part is the "Flap
endonuclease 1 (FEN1)", which describes a function of that structure. The
thirteenth part is the "Summary and perspectives", in which describes
application of this knowledge. sources for this chapters were the excerpts from
two books: "The mechanisms of DNA replication" and "Biochemical
Pathways: An Atlas of Biochemistry and Molecular Biology. Second Edition".of
the reasons why I chose this material for the translation was that the
researching of the molecular mechanism of DNA replication is a central theme of
molecular biology, and now archaeal organisms became popular in the total
genome sequencing age, and most of the DNA replication proteins are now equally
understood by biochemical characterizations.
Now I would like to describe the translation process and the
different translation techniques, that I used. In the first instance, some
words and verbal constructions have the strictly identified value in the field
of biology. There are some simple words (eukaryote - эукариоты, polymerase - полимераза, genom - геном) and some word combinations (domain
of life - домен жизни, pronein factor - протеиновый фактор, initiation factor - фактор инициации). Sometimes while translating a text
I had should to use concretization (Some other protein factors may function
in various archaea, for example a protein that is distantly related to
eukaryotic Cdt1, which plays a crucial role during MCM loading in
Eukaryota, exists in some archaeal organisms, although its function has not
been characterized yet - Некоторые другие белковые факторы могут действовать у различных архей, например белок, отдаленно связаный с эукариотической Cdt1, который играет решающую роль во время комплектации MCM у эукариот, существует у некоторых архей, хотя его функции еще не были охарактеризованы). I also employed
Interpretation when we had to refuse from the literal translation in order to make
the Target Text transparent (A characteristic of the oriC is the
conserved 13 bp repeats, as predicted earlier by bioinformatics, and two of
the repeats are longer and surround apredicted DUE (DNA unwinding element) with
an AT-rich sequence in Pyrococcus genomes - ORIC является сохраненными повторами 13 связывающих белков, как предсказано ранее биоинформатиками, и два из повторов больше и окружают предполагаемый DUE (элемент раскручивания ДНК) с AT-богатых последовательностей в геномах Pyrococcus). used more of the
translation techniques, but describe them here does not seem possible. So, in
the end, I would like to say, that I have got a great experience in
understanding the issues that concern that field of biology. Thus, I not only
improved my biological skills, but also gained the experience needed to improve
my knowledge, as all the latest biological discoveries are always published in
English. And now I have the opportunity to review and analyze the issues
published without waiting for the translation.
English textReplication in Archaea, the Third Domain
of Life
1. Introduction
The accurate duplication and transmission of genetic
information are essential and crucially important for living organisms. The
molecular mechanism of DNA replication has been one of the central themes of
molecular biology, and continuous efforts to elucidate the precise molecular
mechanism of DNA replication have been made since the discovery of the double
helix DNA structure in 1953. The protein factors that function in the DNA
replication process, have been identified to date in the three domains of life
(Figure 1).
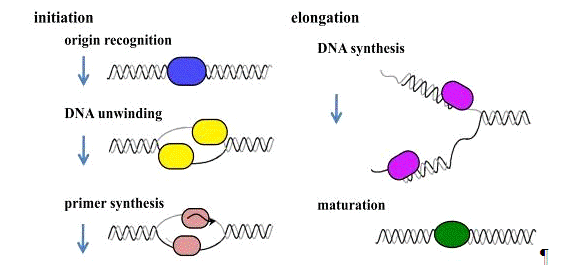
Figure 1. Stage of DNA replication
Archaea, the third domain of life, is a very interesting
living organism to study from the aspects of molecular and evolutional biology.
Rapid progress of whole genome sequence analyses has allowed us to perform
comparative genomic studies. In addition, recent microbial ecology has revealed
that archaeal organisms inhabit not only extreme environments, but also more
ordinary habitats. In these situations, archaeal biology is among the most
exciting of research fields. cells have a unicellular ultrastructure without a
nucleus, resembling bacterial cells, but the proteins involved in the genetic
information processing pathways, including DNA replication, transcription, and
translation, share strong similarities with those of eukaryotes. Therefore,
most of the archaeal proteins were identified as homologues of many eukaryotic
replication proteins, including ORC (origin recognition complex), Cdc6, GINS
(Sld5-Psf1-Psf2-Psf3), MCM (minichromosome maintenance), RPA (replication
protein A), PCNA (proliferating cell nuclear antigen), RFC (replication factor
C), FEN1 (flap endonuclease 1), in addition to the eukaryotic primase, DNA
polymerase, and DNA ligase; these are obviously different from bacterial
proteins and these proteins were biochemically characterized. Their
similarities indicate that the DNA replication machineries of Archaea and
Eukaryota evolved from a common ancestor, which was different from that of
Bacteria. , the archaeal organisms are good models to elucidate the functions
of each component of the eukaryotic type replication machinery complex. Genomic
and comparative genomic research with archaea is made easier by the fact that
the genome size and the number of genes of archaea are much smaller than those
of eukaryotes.
The archaeal replication machinery is probably a simplified
form of that in eukaryotes. On the other hand, it is also interesting that the
circular genome structure is conserved in Bacteria and Archaea and is different
from the linear form of eukaryotic genomes. These features have encouraged us
to study archaeal DNA replication, in the hopes of gaining fundamental insights
into this molecular mechanism and its machinery from an evolutional
perspective. study of bacterial DNA replication at a molecular level started in
about 1960, and then eukaryotic studies followed since 1980. Because Archaea
was recognized as the third domain of life later, the archaeal DNA replication
research became active after 1990. With increasing the available total genome
sequences, the progress of research on archaeal DNA replication has been rapid,
and the depth of our knowledge of archaeal DNA replication has almost caught up
with those of the bacterial and eukaryotic research fields. In this chapter, we
will summarize the current knowledge of DNA replication in Archaea.
2. Replication origin
The basic mechanism of DNA replication was predicted as
“replicon theory” by Jacob et al. They proposed that an initiation factor
recognizes the replicator, now referred to as a replication origin, to start
replication of the chromosomal DNA. Then, the replication origin of E. coli DNA
was identified as oriC (origin of chromosome). The archaeal replication
origin was identified in the Pyrococcus abyssi in 2001 as the first
archaeal replication origin. origin was located just upstream of the gene
encoding the Cdc6 and Orc1-like sequence s in the Pyrococcus genome. We
discovered a gene encoding an amino acid sequence that bore similarity to those
of both eukaryotic Cdc6 and Orc1, which are the eukaryotic initiators. After
confirming that this protein actually binds to the oriC region on the
chromosomal DNA we named the gene product Cdc6/Orc1 due to its roughly equal
homology with regions of eukaryotic Orc1 and Cdc6. The gene consists of an
operon with the gene encoding DNA polymerase D (it was originally called Pol
II, as the second DNA polymerase from Pyrococcus furiosus) in the
genome. characteristic of the oriC is the conserved 13 bp repeats, as
predicted earlier by bioinformatics, and two of the repeats are longer and
surround apredicted DUE (DNA unwinding element) with an AT-rich sequence in Pyrococcus
genomes (Figure 2). The longer repeated sequence was designated as an ORB
(Origin Recognition Box), and it was actually recognized by Cdc6/Orc1 in a Sulfolobus
solfataricus study. The 13 base repeat is called a miniORB, as a minimal
version of ORB. A whole genome microarray analysis of P. abyssi showed
that the Cdc6/Orc1 binds to the oriC region with extremes pecificity,
and the specific binding of the highly purified P. furiosus Cdc6/Orc1 to
ORB and miniORB was confirmed in vitro. It has to be noted that multiple
origins were identified in the Sulfolobus genomes. It is now well
recognized that Sulfolobus has three origins and they work at the same
time in the cell cycle. of the mechanism of how the multiple origins are
utilized for genome replication is an interesting subject in the research field
of archaeal DNA replication. The main questions are how the initiation of
replication from multiple origins is regulated and how the replication forks
progress after the collision of two forks from opposite directions.
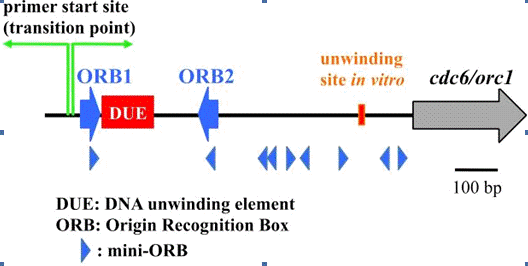
Figure 2. The oriC region in Pyrococcus genome.
3. How does Cdc6/Orc1 recognize oriC?
important step in characterizing the initiation of DNA
replication in Archaea is to understand how the Cdc6/Orc1 protein recognizes
the oriC region. Based upon aminoacid sequence alignments, the archaeal
Cdc6/Orc1 proteins belong to the AAA+ family of proteins. The crystal
structures of the Cdc6/Orc1 protein from Pyrobaculum aerophilum and one
of the two Cdc6/Orc1 proteins, ORC2 from Aeropyrum pernix (the two
homologs in this organism are called ORC1 and ORC2 by the authors) were
determined. These Cdc6/Orc1 proteins consist of three structural domains. I and
II adopt a fold found in the AAA+ family proteins. A winged helix (WH) fold,
which is present in a number of DNA binding proteins, is found in the domain
III. There are four ORBs arranged in pairs on both sides of the DUE in the oriC
region of A. pernix, and ORC1 binds to each ORB as a dimer. A
mechanism was proposed in which ORC1 binds to all four ORBs to introduce a
higher-order assembly for unwinding of the DUE with alterations in both
topology and superhelicity. Furthermore, the crystal structures of S.
solfataricus Cdc6-1 and Cdc6-3 (two of the three Cdc6/Orc1 proteins in this
organism) forming a heterodimer bound to ori2 DNA (one of the three
origins in this organism), and that of A. pernix ORC1 bound to an origin
sequence were determined. These studies revealed that both the N-terminal AAA+
ATPase domain (domain I+II) and C-terminal WH domain (domain III) contribute to
origin DNA binding, and the structural information not only defined the
polarity of initiator assembly on the origin but also indicated the induction
of substantial distortion, which probably triggers the unwinding of the duplex
DNA to start replication, into the DNA strands. These structural data also
provided the detailed interaction mode between the initiator protein and the oriC
DNA. analyses of the Methanothermobactor thermautotrophicus Cdc6-1
protein revealed the essential interaction between an arginine residue
conserved in the archaeal Cdc6/Orc1 and an invariant guanine in the ORB
sequence. P. furiosus Cdc6/Orc1 is difficult to purify in a soluble
form. A specific site in the oriC to start unwinding in vitro,
was identified using the protein prepared by a denaturation-renaturation
procedure recently.
As shown in Figure 2, the local unwinding site is about 670
bp away from the transition site between leading and lagging syntheses, which
was determined earlier by an in vivo replication initiation point (RIP)
assay. Although the details of the replication machinery that must be
established at the unwound site are not fully understood in Archaea, it is
expected to minimally include MCM, GINS, primase, PCNA, DNA polymerase, and
RPA, as described below. The following P. furiosus studies revealed that
the ATPase activity of the Cdc6/Orc1 was completely suppressed by binding to
DNA containing the ORB. proteolysis and DNase I-footprint experiments suggested
that the Cdc6/Orc1 protein changes its conformation on the ORB sequence in the
presence of ATP. The physiological meaning of this conformational change has
not been solved, but it should have an important function to start the
initiation process as in the case of bacterial DnaA protein. In addition,
results from an in vitro recruiting assay indicated that MCM (Mcm
protein complex), the replicative DNA helicase, is recruited onto the oriC region
in a Cdc6/Orc1-dependent, but not ATP-dependent, manner, as described below.
However, this recruitment is not sufficient for the unwinding function of MCM,
and some other function remains to be identified for the functional loading of
this helicase to promote the progression of the DNA replication fork.
4. MCM helicase
unwinding of the oriC region, the replicative helicase
needs to remain loaded to provide continuous unwinding of double stranded DNA
(dsDNA) as the replication forks progress bidirectionally. The MCM protein
complex, consisting of six subunits (Mcm2, 3, 4, 5, 6, and 7), is known to be
the replicative helicase “core” in eukaryotic cells. MCM further interacts with
Cdc45 and GINS, to form a ternary assembly referred to as the “CMG complex”,
that is believed to be the functional helicase in eukaryotic cells (Figure 3).
However, this idea is still not universal for the eukaryotic replicative
helicase.
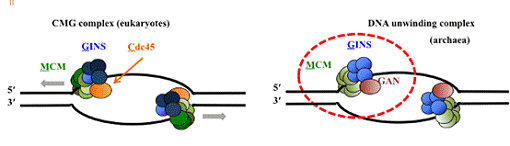
Figure 3. DNA-Unwinding complex in eukaryotes and
archaea.
The CMG complex is the replicative helicase for the template
DNA unwinding reaction in eukaryotes. The archaeal genomes contain the homologs
of the Mcm and Gins proteins, but a Cdc45 homolog has not been identified.
Recent research suggests that a RecJ-like exonuclease GAN, which has weak
sequence homology to that of Cdc45, may work as a helicase complex with MCM and
GINS. archaeal genomes appear to encode at least one Mcm homologue, and the
helicase activities of these proteins from several archaeal organisms have been
confirmed in vitro. In contrast to the eukaryotic MCM, the archaeal
MCMs, consist of a homohexamer or homo double hexamer, having distinct DNA helicase
activity by themselves in vitro, and therefore, these MCMs on their own
may function as the replicative helicase in vivo. structure-function
relationships of the archaeal Mcms have been aggressively studied using
purified proteins and site-directed mutagenesis. An early report using the ChIP
method showed that the P. abyssi Mcm protein preferentially binds to the
origin in vivo in exponentially growing cells. The P. furiosus MCM
helicase does not display significant helicase activity in vitro. However,
the DNA helicase activity was clearly stimulated by the addition of GINS (the
Gins23-Gins51 complex), which is the homolog of the eukaryotic GINS complex
(described below in more detail). This result suggests that MCM works with
other accessory factors to form a core complex in P. furiosus similar to
the eukaryotic CMG complex as described above. archaeal organisms have more
than two Cdc6/Orc1 homologs. It was found that the two Cdc6/Orc1 homologs,
Cdc6-1 and Cdc6-2, both inhibit the helicase activity of MCM in M.
thermautotrophicus. Similarly, Cdc6-1 inhibits MCM activity in S.
solfataricus. In contrast, the Cdc6-2 protein stimulates the helicase
activity of MCM in Thermoplasma acidophilum. Functional interactions
between Cdc6/Orc1 and Mcm proteins need to be investigated in greater detail to
achieve a more comprehensive understanding of the conservation and diversity of
the initiation mechanism in archaeal DNA replication. interesting feature of
DNA replication initiation is that several archaea have multiple genes encoding
Mcm homologs in their genomes. Based on the recent comprehensive genomic
analyses, thirteen archaeal species have more than one mcm gene.
However, many of the mcm genes in the archaeal genomes seem to reside
within mobile elements, originating from viruses. For example, two of the three
genes in the Thermococcus kodakarensis genome are located in regions
where genetic elements have presumably been integrated. establishment of a
genetic manipulation system for T. kodakarensis, is the first for a
hyperthermophilic euryarchaeon, and is advantageous for investigating the
function of these Mcm proteins. Two groups have recently performed gene
disruption experiments for each mcm gene. These experiments revealed
that the knock-out strains for mcm1 and mcm2 were easily
isolated, but mcm3 could not be disrupted. Mcm3 is relatively abundant
in the T. kodakarensis cells. Furthermore, an in vitro experiment
using purified Mcm proteins showed that only Mcm3 forms a stable hexameric
structure in solution. These results support the contention that Mcm3 is the
main helicase core protein in the normal DNA replication process in T.
kodakarensis. functions of the other two Mcm proteins remain to be
elucidated. The genes for Mcm1 and Mcm2 are stably inherited, and their gene
products may perform some important functions in the DNA metabolism in T.
kodakarensis. The DNA helicase activity of the recombinant Mcm1 protein is
strong in vitro, and a distinct amount of the Mcm1 protein is present in
T. kodakarensis cells. Moreover, Mcm1 functionally interacts with the
GINS complex from T. kodakarensis. These observations strongly suggest
that Mcm1 does participate in some aspect of DNA transactions, and may be
substituted with Mcm3. immunoprecipitation experiments showed that Mcm1
co-precipitated with Mcm3 and GINS, although they did not form a
heterohexameric complex, suggesting that Mcm1 is involved in the replisome or
repairsome and shares some function in T. kodakarensis cells. Although
western blot analysis could not detect Mcm2 in the extract from exponentially
growing T. kodakarensis cells, a RT-PCR experiment detected the
transcript of the mcm2 gene in the cells (Ishino et al., unpublished).
The recombinant Mcm2 protein also has ATPase and helicase activities in
vitro. Therefore, the mcm2 gene is expressed under normal growth
conditions and may work in some process with a rapid turnover. Further
experiments to measure the efficiency of mcm2 gene transcription by
quantitative PCR, as well as to assess the stability of the Mcm2 protein in the
cell extract, are needed. analyses investigating the sensitivities of the Δmcm1 and Δmcm2 mutant strains to DNA
damage caused by various mutagens, as reported for other DNA repair-related
genes in T. kodakarensis, may provide a clue to elucidate the functions
of these Mcm proteins. Methanococcus maripaludis S2 harbors four mcm genes
in its genome, three of which seem to be derived from phage, a shotgun
proteomics study detected peptides originating from three out of the four mcm
gene products. Furthermore, the four gene products co-expressed in E.
coli cells were co-purified in the same fraction. These results suggest
that multiple Mcm proteins are functional in the M. maripaludis cells.
5. Recruitment of Mcm to the oriC region
important question is how MCM is recruited onto the unwound
region of oriC. The detailed loading mechanism of the MCM helicase has not been
elucidated. It is believed that archaea utilize divergent mechanisms of MCM
helicase assembly at the oriC. in vitro recruiting assay showed that P.
furiosus MCM is recruited to the oriC DNA in a Cdc6/Orc1-dependent manner. This
assay revealed that preloading Cdc6/Orc1 onto the ORB DNA resulted in a clear
reduction in MCM recruitment to the oriC region, suggesting that free Cdc6/Orc1
is preferable as a helicase recruiter, to associate with MCM and bring it to
oriC. It would be interesting to understand how the two tasks, origin
recognition and MCM recruiting, are performed by the Cdc6/Orc1 protein, because
the WH domain, which primarily recognizes and binds ORB, also has strong
affinity for the Mcm protein. assembly of the Mcm protein onto the ORB DNA by
the Walker A-motif mutant of P. furiosus Cdc6/Orc1 occurred with the
same efficiency as the wild type Cdc6/Orc1. The DNA binding of P. furiosus Cdc6/Orc1
was not drastically different in the presence and absence of ATP, as in the
case of the initiator proteins from Archaeoglobus fulgidus, S.
solfataricus, and A. pernix. Therefore, it is still not known whether
the ATP binding and hydrolysis activity of Cdc6/Orc1 regulates the Mcm protein
recruitment onto oriC in the cells. more important issue is the very low
efficiency of the Mcm protein recruitment in the reported in vitro assay.
Quantification of the recruited Mcm protein by the in vitro assay showed
that less than one Mcm hexamer was recruited to the ORB. The linear DNA
containing ORB1 and ORB2, used in the recruiting assay, may not be suitable to
reconstitute the archaeal DNA replication machinery and a template that more
closely mimics the chromosomal DNA may be required. , it may be that as yet
unidentified proteins are required to achieve efficient in vitro helicase
loading in the P. furiosus cells. Finally, it will ultimately be
necessary to construct a more defined in vitro replication system to
analyze the regulatory functions of Cdc6/Orc1 precisely during replication
initiation. M. thermautotrophicus, the Cdc6-2 proteins can dissociate
the Mcm multimers. The activity of Cdc6-2 might be required as the MCM helicase
loader in this organism. The interaction between Cdc6/Orc1 and Mcm is probably
general. However, the effect of Cdc6/Orc1 on the MCM helicase activity differs
among various organisms, as described above. Some other protein factors may
function in various archaea, for example a protein that is distantly related to
eukaryotic Cdt1, which plays a crucial role during MCM loading in Eukaryota,
exists in some archaeal organisms, although its function has not been
characterized yet.
6. GINS
The eukaryotic GINS complex was originally identified in Saccharomyces
cerevisiae as essential protein factor for the initiation of DNA
replication. GINS consists of four different proteins, Sld5, Psf1, Psf2, and
Psf3 (therefore, GINS is an acronym for Japanese go-ichi-ni-san, meaning
5-1-2-3, after these four subunits). The amino acid sequences of the four
subunits in the GINS complex share some conservation, suggesting that they are
ancestral paralogs. However, most of the archaeal genomes have only one gene encoding
this family protein, and more interestingly, the Crenarchaeota and
Euryarchaeota (the two major subdomains of Archaea) characteristically have two
genes with sequences similar to Psf2 and Psf3, and Sld5 and Psf1, respectively
referred to as Gins23 and Gins51. Gins homolog, designated as Gins23, was
biochemically detected in S. solfataricus as the first Gins protein in
Archaea, in a yeast two-hybrid screening for interaction partners of the Mcm
protein, and another subunit, designated as Gins15, was identified by
mass-spectrometry analysis of an immunoaffinity-purified native GINS from an S.
solfataricus cell extract. The S. solfataricus GINS, composed of two
proteins, Gins23 and Gins15, forms a tetrameric structure with a 2:2 molar
ratio. The GINS from P. furiosus, a complex of Gins23 and Gins51 with a
2:2 ratio, was identified as the first euryarchaeal GINS. Gins51 was preferred
over Gins15 because of the order of the name of GINS. MCM2-7 hexamer was
copurified in complex with Cdc45 and GINS from Drosophila melanogaster embryo
extracts and S. cerevisiae lysates, and the “CMG (Cdc45-MCM2-7-GINS)
complex” (Figure 3), as described above, should be important for the function
of the replicative helicase. The CMG complex was also associated with the
replication fork in Xenopus laevis egg extracts, and a large molecular
machine, containing Cdc45, GINS, and MCM2-7, was proposed as the unwindosome to
separate the DNA strands at the replication fork. , GINS must be a critical
factor for not only the initiation process, but also the elongation process in
eukaryotic DNA replication. S. solfataricus GINS interacts with MCM and
primase, suggesting that GINS is involved in the replisome. The concrete
function of GINS in the replisome remains to be determined. No stimulation or
inhibition of either the helicase or primase activity was observed by the
interaction with S. solfataricus GINS in vitro. On the other
hand, the DNA helicase activity of P. furiosus MCM is clearly stimulated
by the addition of the P. furiosus GINS complex, as described above.
contrast to S. solfataricus and P. furiosus, which each express a
Gins23 and Gins51, Thermoplasma acidophilum has a single Gins homolog,
Gins51. The recombinant Gins51 protein from T. acidophilum was confirmed
to form a homotetramer by gel filtration and electron microscopy analyses.
Furthermore, a physical interaction between T. acidophilum Gins51 and
Mcm was detected by a surface plasmon resonance analysis (SPR). Although the T.
acidophilum Gins51 did not affect the helicase activity of its cognate MCM,
when the equal ratio of each molecule was tested in vitro, an excess
amount of Gins51 clearly stimulated the helicase activity (Ogino et al.,
unpublished). In the case of T. kodakarensis, the ATPase and helicase
activities of MCM1 and MCM3 were clearly stimulated by T. kodakarensis GINS
in vitro. It is interesting that the helicase activity of MCM1 was
stimulated more than that of MCM3. interactions between the T. kodakarensis Gins
and Mcm proteins were also detected. These reports suggested that the MCM-GINS
complex is a common part of the replicative helicase in Archaea (Figure 3).
Recently, the crystal structure of the T. kodakarensis GINS tetramer,
composed of Gins51 and Gins23 was determined, and the structure was conserved
with the reported human GINS structures. Each subunit of human GINS shares a
similar fold, and assembles into the heterotetramer of a unique trapezoidal
shape. Sld5 and Psf1 possess the α-helical (A) domain at the
N-terminus and the β-stranded domain (B) at the
C-terminus (AB-type). the other hand, Psf2 and Psf3 are the permuted version
(BA-type). The backbone structure of each subunit and the tetrameric assembly
of T. kodakarensis GINS are similar to those of human GINS. However, the
location of the C-terminal B domain of Gins51 is remarkably different between
the two GINS structures. homology model of the homotetrameric GINS from T.
acidophilum was performed using the T. kodakarensis GINS crystal
structure as a template. The Gins 51 protein has a long disordered region
inserted between the A and B domains and this allows the conformation of the
C-terminal domains to be more flexible. This domain arrangement leads to the
formation of an asymmetric homotetramer, rather than a symmetrical assembly, of
the T. kodakarensis GINS.Cdc45 protein is ubiquitously distributed from
yeast to human, supporting the notion that the formation of the CMG complex is
universal in the eukaryotic DNA replication process. However, no archaeal
homologue of Cdc45 has been identified. A recent report of bioinformatic
analysis showed that the primary structure of eukaryotic Cdc45 and prokaryotic
RecJ share a common ancestry. Indeed, a homolog of the DNA binding domain of
RecJ has been co-purified with GINS from S. solfataricus. experiment
detected the stimulation of the 5’-3’ exonuclease activity of the RecJ homologs
from P. furiosus and T. kodakarensis by the cognate GINS
complexes (Ishino et al., unpublished). The RecJ homolog from T.
kodakarensis forms a stable complex with the GINS, and the 5’-3’
exonuclease activity is enhanced in vitro; therefore, the RecJ homolog
was designated as GAN, from GINS-Associated Nuclease in a very recent paper.
related report found that the human Cdc45 structure obtained by the small angle
X-ray scattering analysis (SAXS) is consistent with the crystallographic
structure of the RecJ family members. These current findings will promote
further research on the structures and functions of the higher-order
unwindosome in archaeal and eukaryotic cells.
7. Primase
initiate DNA strand synthesis, a primase is required for the
synthesis of a short oligonucleotide, as a primer. The DnaG and p48-p58
proteins are the primases in Bacteria and Eukaryota, respectively. The p48-p58
primase is further complexed with p180 and p70, to form DNA polymerase α-primase complex. The
catalytic subunits of the eukaryotic (p48) and archaeal primases, share a
little, but distinct sequence homology with those of the family X DNA
polymerases. first archaeal primase was identified from Methanococcus
jannaschii, as an ORF with a sequence similar to that of the eukaryotic
p48. The gene product exhibited DNA polymerase activity and was able to
synthesize oligonucleotides on the template DNA. characterized the p48-like
protein (p41) from P. furiosus. Unexpectedly, the archaeal p41 protein
did not synthesize short RNA by itself, but preferentially utilized
deoxynucleotides to synthesize DNA strands up to several kilobases in length.
Furthermore, the gene neighboring the p41 gene encodes a protein with very weak
similarity to the p58 subunit of the eukaryotic primase. The gene product,
designated p46, actually forms a stable complex with p41, and the complex can
synthesize a short RNA primer, as well as DNA strands of several hundred
nucleotides in vitro. short RNA but not DNA primers were identified in Pyrococcus
cells, and therefore, some mechanism to dominantly use RNA primers exists
in the cells. research on the primase homologs from S. solfataricus, Pyrococcus
horikoshii, and P. abyssi showed similar properties in vitro.
Notably, p41 is the catalytic subunit, and the large one modulates the activity
in the heterodimeric archaeal primases. small and large subunits are also
called PriS and PriL, respectively. The crystal structure of the N-terminal
domain of PriL complexed with PriS of S. solfataricus primase revealed
that PriL does not directly contact the active site of PriS, and therefore, the
large subunit may interact with the synthesized primer, to adjust its length to
a 7-14 mer. The structure of the catalytic center is similar to those of the
family X DNA polymerases. 3’-terminal nucleotidyl transferase activity,
detected in the S. solfataricus primase, and the gap-filling and
strand-displacement activities in the P. abyssi primase also support the
structural similarity between PriS and the family X DNA polymerases. A unique
activity, named PADT (template-dependent Polymerization Across Discontinuous
Template), in the S. solfataricus PriSL complex was published very
recently. activity may be involved in double-strand break repair in Archaea.
The archaeal genomes also encode a sequence similar to the bacterial type DnaG
primase. The DnaG homolog from the P. furiosus genome was expressed in E.
coli, but the protein did not show any primer synthesis activity in
vitro, and thus the archaeal DnaG-like protein may not act as a primase in Pyrococcus
cells. DnaG-like protein was shown to participate in RNA degradation, as an
exosome component. However, a recent paper reported that a DnaG homolog from S.
solfataricus actually synthesizes primers with a 13 nucleotide length. It
would be interesting to investigate if the two different primases share the
primer synthesis for leading and lagging strand replication, respectively, in
the Sulfolobus cells, as the authors suggested. proposed hypothesis
about the evolution of PriSL and DnaG from the last universal common ancestor
(LUCA) is interesting. The Sulfolobus PriSL protein was shown to
interact with Mcm through Gins23. This primase- helicase interaction probably
ensures the coupling of DNA unwinding and priming during the replication fork
progression. , the direct interaction between PriSL and the clamp loader RFC
(described below) in S. solfataricus may regulate the primer synthesis
and its transfer to DNA polymerase in archaeal cells.
8. Single-stranded DNA binding protein
single-stranded DNA binding protein, which is called SSB in
Bacteria and RPA in Archaea and Eukaryota, is an important factor to protect
the unwound single-stranded DNA from nuclease attack, chemical modification,
and other disruptions during the DNA replication and repair processes. SSB and
RPA have a structurally similar domain containing a common fold, called the OB
(oligonucleotide/oligosaccharide binding)-fold, although there is little amino
acid sequence similarity between them.common structure suggests that the
mechanism of single-stranded DNA binding is conserved in living organisms
despite the lack of sequence similarity. E. coli SSB is a homotetramer
of a 20 kDa peptide with one OB-fold, and the SSBs from Deinococcus
radiodurans and Thermus aquaticus consist of a homodimer of the
peptide containing two OB-folds. eukaryotic RPA is a stable heterotrimer,
composed of 70, 32, and 14 kDa proteins. RPA70 contains two tandem repeats of
an OB-fold, which are responsible for the major interaction with a
single-stranded DNA in its central region. The N-terminal and C-terminal
regions of RPA70 mediate interactions with RPA32 and also with many cellular or
viral proteins. RPA32 contains an OB-fold in the central region, and the
C-terminal region interacts with other RPA subunits and various cellular
proteins. RPA14 also contains an OB-fold. eukaryotic RPA interacts with the
SV40 T-antigen and the DNA polymerase α-primase complex, and thus
forms part of the initiation complex at the replication origin. The RPA also
stimulates Polα-primase activity and PCNA-dependent Pol δ activity. The RPAs from M.
jannaschii and M. thermautotrophicus were reported in 1998, as the
first archaeal single-stranded DNA binding proteins. These proteins share amino
acid sequence similarity with the eukaryotic RPA70, and contain four or five
repeated OB-fold and one zincfinger motif. M. jannaschii RPA exists as a
monomer in solution, and has single-strand DNA binding activity. On the other
hand, P. furiosus RPA forms a complex consisting of three distinct
subunits, RPA41, RPA32, and RPA14, similar to the eukaryotic RPA. The P. furiosus
RPA strikingly stimulates the RadA-promoted strand-exchange reaction in
vitro. the euryarchaeal organisms have a eukaryotic-type RPA homologue, the
crenarchaeal SSB proteins appear to be much more related to the bacterial
proteins, with a single OB fold and a flexible C-terminal tail. However, the
crystal structure of the SSB protein from S. solfataricus showed that
the OB-fold domain is more similar to that of the eukaryotic RPAs, supporting
the close relationship between Archaea and Eukaryota. RPA from Methanosarcina
acetivorans displays a unique property. Unlike the multiple RPA proteins
found in other archaea and eukaryotes, each subunit of the M. acetivorans RPAs,
RPA1, RPA2, and RPA3, have 4, 2, and 2 OB-folds, respectively, and can act as a
distinct single-stranded DNA-binding proteins. Furthermore, each of the three
RPA proteins, as well as their combinations, clearly stimulates the primer
extension activity of M. acetivorans DNA polymerase BI in vitro,
as shown previously for bacterial SSB and eukaryotic RPA. of SSB and RPA
suggested that they are composed of different combinations of the OB fold.
Bacterial and eukaryotic organisms contain one type of SSB or RPA,
respectively. In contrast, archaeal organisms have various RPAs, composed of
different organizations of OB-folds. A hypothesis that homologous recombination
might play an important role in generating this diversity of OB-folds in
archaeal cells was proposed, based on experiments characterizing the engineered
RPAs with various OB-folds.
9. DNA polymerase
polymerase catalyzes phosphodiester bond formation between
the terminal 3’-OH of the primer and the α-phosphate of the incoming
triphosphate to extend the short primer, and is therefore the main player of
the DNA replication process. Based on the amino acid sequence similarity, DNA
polymerases have been classified into seven families, A, B, C, D, E, X, and Y.
fundamental ability of DNA polymerases to synthesize a deoxyribonucleotide
chain is widely conserved, but more specific properties, including
processivity, synthesis accuracy, and substrate nucleotide selectivity, differ
depending on the family. The enzymes within the same family have basically
similar properties. E. coli has five DNA polymerases, and Pol I, Pol II,
and Pol III belong to families A, B, and C, respectively. Pol IV and Pol V are
classified in family Y, as the DNA polymerases for translesion synthesis (TLS).
In eukaryotes, the replicative DNA polymerases, Pol α, Pol δ, and Pol ε, belong to family B, and
the translesion DNA polymerases, η, ι, and κ, belong to family Y. most
interesting feature discovered at the inception of this research area was that
the archaea indeed have the eukaryotic Pol α-like (Family B) DNA
polymerases. Members of the Crenarchaeota have at least two family B DNA
polymerases . On the other hand, there is only one family B DNA polymerase in
the Euryarchaeota. Instead, the euryarchaeal genomes encode a family D DNA
polymerase, proposed as Pol D, which seems to be specific for these archaeal
organisms and has never been found in other domains. genes for family Y-like
DNA polymerases are conserved in several, but not all, archaeal genomes. The
role of each DNA polymerase in the archaeal cells is still not known, although
the distribution of the DNA polymerases is getting clearer. first family D DNA
polymerase was identified from P. furiosus, by screening for DNA
polymerase activity in the cell extract. The corresponding gene was cloned,
revealing that this new DNA polymerase consists of two proteins, named DP1 and
DP2, and that the deduced amino acid sequences of these proteins were not
conserved in the DNA polymerase families. P. furiosus Pol D exhibits
efficient strand extension activity and strong proofreading activity. family D
DNA polymerases were also characterized by several groups. The Pol D genes had
been found only in Euryarchaeota. However, recent environmental genomics and
cultivation efforts revealed novel phyla in Archaea: Thaumarchaeota,
Korarchaeota, and Aigarchaeota, and their genome sequences harbor the genes
encoding Pol D. A genetic study on Halobacterium sp. NRC-1 showed that
both Pol B and Pol D are essential for viability. interesting issue is to
elucidate whether Pol B and Pol D work together at the replication fork for the
synthesis of the leading and lagging strands, respectively. According to the
usage of an RNA primer and the presence of strand displacement activity, Pol D
may catalyze lagging strand synthesis.
Thaumarchaeota and Aigarchaeota harbor the genes encoding Pol
D and crenarchaeal Pol BII, while Korarchaeota encodes Pol BI, Pol BII and Pol
D. Biochemical characterization of these gene products will contribute to
research on the evolution of DNA polymerases in living organisms. hypothesis
that the archaeal ancestor of eukaryotes encoded three DNA polymerases, two
distinct family B DNA polymerases and a family D DNA polymerase, which all
contributed to the evolution of the eukaryotic replication machinery,
consisting of Pol α, δ, and ε, has been proposed. A
protein is encoded in the plasmid pRN1 isolated from a Sulfolobus strain.
This protein, ORF904 (named RepA), has primase and DNA polymerase activities in
the N-terminal domain and helicase activity in the C-terminal domain, and is
likely to be essential for the replication of pRN1. amino acid sequence of the
N-terminal domain lacks homology to any known DNA polymerases or primases, and
therefore, family E is proposed. Similar proteins are encoded by various
archaeal and bacterial plasmids, as well as by some bacterial viruses. , one
protein, tn2-12p, encoded in the plasmid pTN2 isolated from Thermococcus
nautilus, was experimentally identified as a DNA polymerase in this family.
This enzyme is likely responsible for the replication of the plasmids. Further
investigations of this family of DNA polymerases will be interesting from an
evolutional perspective.
10. PCNA and RFC
sliding clamp with the doughnut-shaped ring structure is
conserved among living organisms, and functions as a platform or scaffold for
proteins to work on the DNA strands. The eukaryotic and archaeal PCNAs form a
homotrimeric ring structure, which encircles the DNA strand and anchors many
important proteins involved in DNA replication and repair (Figure 4). works as
a processivity factor that retains the DNA polymerase on the DNA by binding it
on one surface (front side) of the ring for continuous DNA strand synthesis in
DNA replication (Figure 5). To introduce the DNA strand into the central hole
of the clamp ring, a clamp loader is required to interact with the clamp and
open its ring. The archaeal and eukaryotic clamp loader is called RFC (Figure
5). most studied archaeal PCNA and RFC molecules to date are P. furiosus PCNA
and RFC. The PCNA and RFC molecules are essential for DNA polymerase to perform
processive DNA synthesis. The molecular mechanism of the clamp loading process
has been actively investigated (Figure 5). intermediate PCNA-RFC-DNA complex,
in which the PCNA ring is opened with out-of plane mode, was detected by a
single particle analysis of electron microscopic images using P. furiosus proteins
(Figure 6). crystal structure of the complex, including the ATP-bound clamp
loader, the ring-opened clamp, and the template-primer DNA, using proteins from
bacteriophage T4, has recently been published, and our knowledge about the
clamp loading mechanism is continuously progressing.
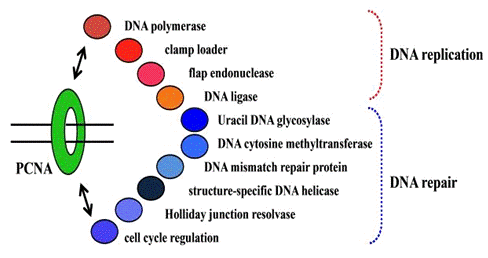
Figure 4. PCNA-interacting proteins.
After clamp loading, DNA polymerase accesses the clamp and
the polymerase-clamp complex performs processive DNA synthesis. Therefore,
structural and functional analyses of the DNA polymerase-PCNA complex is the
next target to elucidate the overall mechanisms of replication fork
progression. The PCNA interacting proteins contain a small conserved sequence
motif, called the PIP box, which binds to a common site on PCNA. The PIP box
consists of the sequence “Qxxhxxaa”, where “x” represents any amino acid, “h”
represents a hydrophobic residue (e.g. L, I or M), and “a” represents an
aromatic residue (e.g. F, Y or W). Archaeal DNA polymerases have PIP box-like
motifs in their sequences. However, only a few studies have experimentally
investigated the function of the motifs. The crystal structure of P.
furiosus Pol B complexed with a monomeric PCNA mutant was determined, and a
convincing model of the polymerase-PCNA ring interaction was constructed. This
study revealed that a novel interaction is formed between a stretched loop of
PCNA and the thumb domain of Pol B, in addition to the authentic PIP box.
comparison of the model structure with the previously reported structures of a
family B DNA polymerase from RB69 phage, complexed with DNA, suggested that the
second interaction site plays a crucial role in switching between the
polymerase and exonuclease modes, by inducing a PCNA-polymerase complex
configuration that favors synthesis over editing. putative mechanism for the
fidelity control of replicative DNA polymerases is supported by experiments, in
which mutations at the second interaction site enhanced the exonuclease
activity in the presence of PCNA. Furthermore, the three-dimensional structure
of the DNA polymerase-PCNA-DNA ternary complex was analyzed by electron
microscopic (EM) single particle analysis. structural view revealed the entire
domain configuration of the trimeric ring of PCNA and DNA polymerase, including
the protein-protein or protein-DNA contacts. This architecture provides clearer
insights into the switching mechanism between the editing and synthesis modes.

Figure 5. Mechanisms of processive DNA synthesis
In contrast to most euryarchaeal organisms, which have a
single PCNA homolog forming a homotrimeric ring structure, the majority of
crenarchaea have multiple PCNA homologues, and they are capable of forming
heterotrimeric rings for their functions. It is especially interesting that the
three PCNAs, PCNA1, PCNA2, and PCNA3, specifically bind PCNA binding proteins,
including DNA polymerases, DNA ligases, and FEN-1 endonuclease. structural
studies of the heterologous PCNA from S. solfataricus revealed that the
interaction modes between the subunits are conserved with those of the
homotrimeric PCNAs. T. kodakarensis is the only euryarchaeal species
that has two genes encoding PCNA homologs on the genome. These two genes from
the T. kodakarensis genome, and the highly purified gene products, PCNA1
and PCNA2, were characterized. stimulated the DNA synthesis reactions of the
two DNA polymerases, Pol B and Pol D, from T. kodakarensis in vitro.
PCNA2 however only had an effect on Pol B. The T. kodakarensis strain
with pcna2 disruption was isolated, whereas gene disruption for pcna1
was not possible. These results suggested that PCNA1 is essential for DNA
replication, and PCNA2 may play a different role in T. kodakarensis cells.
sensitivities of the Δpcna2 mutant strain to ultraviolet
irradiation (UV), methyl methanesulfonate (MMS) and mitomycin C (MMC) were
indistinguishable to those of the wild type strain. Both PCNA1 and PCNA2 form a
stable ring structure and work as a processivity factor for T. kodakarensis Pol
B in vitro. The crystal structures of the two PCNAs revealed the
different interactions at the subunit-subunit interfaces. the other hand, the
archaeal RFC consists of two proteins, RFCS (small) and RFCL (large), in a 4 to
1 ratio. A different form of RFC, consisting of three subunits, RFCS1, RFCS2,
and RFCL, in a 3 to 1 to 1 ratio, was also identified from M. acetivorans. The
three subunits of RFC may represent an intermediate stage in the evolution of
the more complex RFC in Eukaryota from the less complex RFC in Archaea.
Figure 6. Electron Microscopic Analysis of P.
furious DNA polymerase-PCNA-DNA complex.
subunit organization and the spatial distribution of the
subunits in the M. acetivorans RFC complex were analyzed and compared
with those of the E. coli γ-complex, which is also a
pentamer consisting of three different proteins. These two clamp loaders adopt
similar subunit organizations and spatial distributions, but the functions of
the individual subunits are likely to be diverse.
11. DNA ligase
ligase is essential to connect the Okazaki fragments of the
discontinuous strand synthesis during DNA replication, and therefore, it
universally exists in all living organisms. This enzyme catalyzes
phosphodiester bond formation via three nucleotidyl transfer steps. In the
first step, DNA ligase forms a covalent enzyme-AMP intermediate, by reacting
with ATP or NAD+ as a cofactor. In the second step, DNA ligase recognizes the
substrate DNA, and the AMP is subsequently transferred from the ligase to the
5’-phosphate terminus of the DNA, to form a DNA-adenylate intermediate
(AppDNA). In the final step, the 5’-AppDNA is attacked by the adjacent
3’-hydroxy group of the DNA and a phosphodiester bond is formed. DNA ligases
are grouped into two families, according to their requirement for ATP or NAD+
as a nucleotide cofactor in the first step reaction. ATPdependent DNA ligases
are widely found in all three domains of life, whereas NAD+-dependent DNA
ligases exist mostly in Bacteria. Some halophilic archaea and eukaryotic
viruses also have NAD+-dependent enzymes. genes (LIG1, LIG3 and LIG4)
encoding ATP-dependent DNA ligases have been identified in the human genome to
date and DNA ligase I (Lig I), encoded by LIG1, is a replicative enzyme
that joins Okazaki fragments during DNA replication. The first gene encoding a
eukaryotic-like ATP-dependent DNA ligase was found in the thermophilic
archaeon, Desulfolobus ambivalens. Subsequent identifications of the DNA
ligases from archaeal organisms revealed that these enzymes primarily use ATP
as a cofactor. However, this classification may not be so strict. The
utilization of NAD+, as well as ATP, as a cofactor has been observed in several
DNA ligases, including those from T. kodakarensis, T. fumicolans, P.
abyssi, Thermococcus sp. NA1, T. acidophilum, Picrophilus
torridus, and Ferroplasma acidophilum, although ATP is evidently
preferable in all of the cases. dual co-factor specificity (ATP/NAD+) is an
interesting feature of these DNA ligase enzymes and it will be enlightening to
investigate the structural basis for this. Another dual co-factor specificity
exists in the archaeal DNA ligases, which use ADP as well as ATP, as found in
the enzymes from A. pernix and Staphylothermus marinus, and in
the case of Sulfobococcus zilligii, GTP is also the functional cofactor.
The DNA ligases from P. horikoshii and P. furiosus have a strict
ATP preference. biochemical data have not been obtained to resolve the issue of
dual co-factor specificity, and further biochemical and structural analyses are
required. The crystal structure of P. furiosus DNA ligase was solved and
the physical and functional interactions between the DNA ligase and PCNA was
shown. The detailed interaction mode between human Lig I and PCNA is somewhat
unclear, because of several controversial reports. stimulatory effect of P.
furiosus PCNA on the enzyme activity of the cognate DNA ligase was observed
at a high salt concentration, at which a DNA ligase alone cannot bind to a
nicked DNA substrate. Interestingly, the PCNA-binding site is located in the
middle of the N-terminal DNA binding domain (DBD) of the P. furiosus DNA
ligase, and the binding motif, QKSFF, which is proposed as a shorter version of
the PIP box, is actually looped out from the protein surface. Interestingly,
this motif is located in the middle of the protein chain, rather than the N- or
C-terminal region, where the PIP boxes are usually located. To confirm that
this motif is conserved in the archaeal/eukaryotic DNA ligases, the physical
and functional interactions between A. pernix DNA ligase and PCNA was
analyzed and the interaction was shown to mainly depend on the phenylalanine
132 residue, which is located in the predicted region from the multiple
sequence alignment of the ATP-dependent DNA ligases. crystal structure of the
human Lig I, complexed with DNA, was solved as the first ATPdependent mammalian
DNA ligase, although the ligase was an N-terminal truncated form. The structure
comprises the N-terminal DNA binding domain, the middle adenylation domain, and
the C-terminal OB-fold domain. The crystal structure of Lig I (residues 233 to
919) in complex with a nicked, 5'-adenylated DNA intermediate revealed that the
enzyme redirects the path of the dsDNA, to expose the nick termini for the
strand-joining reaction. The N-terminal DNA-binding domain works to encircle
the DNA substrate like PCNA and to stabilize the DNA in a distorted structure,
positioning the catalytic core on the nick. crystal structure of the full
length DNA ligase from P. furiosus revealed that the architecture of
each domain resembles those of Lig I, but the domain arrangements strikingly
differ between the two enzymes. This domain rearrangement is probably derived
from the “domain-connecting” role of the helical extension conserved at the
C-termini in the archaeal and eukaryotic DNA ligases. The DNA substrate in the
open form of Lig I is replaced by motif VI at the C-terminus, in the closed
form of P. furiosus DNA ligase. the shapes and electrostatic
distributions are similar between motif VI and the DNA substrate, suggesting
that motif VI in the closed state mimics the incoming substrate DNA. The
subsequently solved crystal structure of S. solfataricus DNA ligase is
the fully open structure, in which the three domains are highly extended. In
this work, the S. solfataricus ligase-PCNA complex was also analyzed by
SAXS. S. solfataricus DNA ligase bound to the PCNA ring still retains an
open, extended conformation. The closed, ring-shaped conformation observed in
the Lig I structure as described above is probably the active form to catalyze
a DNA end-joining reaction, and therefore, it is proposed that the
open-to-closed movement occurs for ligation, and the switch in the
conformational change is accommodated by a malleable interface with PCNA, which
serves as an efficient platform for DNA ligation. the publication of these
crystal structures, the three-dimensional structure of the ternary complex,
consisting of DNA ligase-PCNA-DNA, using the P. furiosus proteins was
obtained by EM single particle analysis. In the complex structure, the three
domains of the crescent-shaped P. furiosus DNA ligase surround the
central DNA duplex, encircled by the closed PCNA ring. The relative
orientations of the ligase domains remarkably differ from those of the crystal
structures, and therefore, a large domain rearrangement occurs upon ternary
complex formation. the EM image model, the DNA ligase contacts PCNA at two
sites, the conventional PIP box and a novel second contact in the middle
adenylation domain. It is also interesting that a substantial DNA tilt from the
PCNA ring axis is observed. Based on these structural analyses, a mechanism in
which the PCNA binding proteins are bound and released sequentially. In fact,
most of the PCNA binding proteins share the same binding sites in the
interdomain connecting loop (IDCL) and the C-terminal tail of the PCNA. The
structural features exclude the possibility that the three proteins contact the
single PCNA ring simultaneously, because DNA ligase occupies two of the three
subunits of the PCNA trimer. the case of the RFCPCNA-DNA complex structure
obtained by the same EM technique, RFC entirely covers the PCNA ring, thus
blocking the access of other proteins. These ternary complexes appear to favor
a mechanism involving the sequential binding and release of replication
factors.
12. Flap endonuclease 1 (FEN1)
processing of Okazaki fragments to make a continuous DNA
strand is essential for the lagging strand synthesis in asymmetric DNA
replication. The primase-synthesized RNA/DNA primers need to be removed to join
the Okazaki fragments into an intact continuous strand DNA. Flap endonuclease 1
(FEN1) is mainly responsible for this task. Okazaki fragment maturation is
highly coordinated with continuous DNA synthesis, and the interactions of DNA
polymerase, FEN1, and DNA ligase with PCNA allow these enzymes to act
sequentially during the maturation process, as described above. FEN1, a
structure-specific 5’-endonuclease, specifically recognizes a dsDNA with an
unannealed 5’-flap. the eukaryotic Okazaki fragment processing system, 5’-flap
DNA structures are formed by the strand displacement activity of DNA polymerase
δ. Lig I seals the nick
after the flapped DNA is cleaved by FEN1. These processing steps are
facilitated by PCNA. The interactions between eukaryotic FEN1 and PCNA have
been well characterized, and the stimulatory effect of PCNA on the FEN1
activity was also shown. crystal structure of the human FEN1-PCNA complex
revealed three FEN1 molecules bound to each PCNA subunit of the trimer ring in
different configurations. Based on these structural analyses together with the
description in the DNA ligase section, a flip-flop transition mechanism, which
enables proteins to internally switch for different functions on the same DNA
clamp are currently being considered. The eukaryotic homologs of FEN1 were
found in Archaea. crystal structures of FEN1 from M. jannaschii, P. furiosus,
P. horikoshii, A. fulgidus, and S. solfataricus have been
determined. In addition, detailed biochemical studies were performed on P.
horikoshii FEN1. Thus, studies of the archaeal FEN1 proteins have provided
important insights into the structural basis of the cleavage reaction of the
flapped DNA. Our recent research showed that the flap endonuclease activity of P.
furiosus FEN1 was stimulated by PCNA. , the stimulatory effect of PCNA on
the sequential action of FEN1 and DNA ligase was observed in vitro (Kiyonari
et al., unpublished). Based on these results, a model of the molecular
switching mechanisms of the last steps of Okazaki-fragment maturation was
constructed. The quaternary complex of FEN1-Lig-PCNA-DNA was also isolated for
the EM single particle analysis. These studies will provide more concrete image
of the molecular mechanism.
13. Summary and perspectives
on the molecular mechanism of DNA replication has been a
central theme of molecular biology. Archaeal organisms became popular in the
total genome sequencing age, as described above, and most of the DNA
replication proteins are now equally understood by biochemical
characterizations. In addition, the archaeal studies are especially interesting
to understand the mechanisms by which cells live in extreme environmental
conditions. , it is also noteworthy that the proteins from the
hyperthermophilic archaea are more stable than those from mesophilic organisms,
and they are advantageous for the structural and functional analyses of higher-ordered
complexes, such as the replisome. Studies on the higher-ordered complexes,
rather than single proteins, are essential for understanding each of the events
involved in DNA metabolism, and the archaeal research will continuously
contribute to the development and advancement of the DNA replication research
field, as summarized in part in a recent review. addition to basic molecular
biology research, DNA replication proteins from thermophiles have been quite
useful reagents for gene manipulations, including genetic diagnosis, forensic
DNA typing, and detection of bacterial and virus infections, as well as basic
research. Numerous enzymes have been commercialized around the world, and are
utilized daily. An example of the successful engineering of an archaeal DNA
polymerase for PCR is the creation of the fusion protein between P. furiosus
Pol B and a nonspecific dsDNA binding protein, Sso7d, from S.
solfataricus, by genetic engineering techniques. fusion DNA polymerase
overcame the low processivity of the wild type Pol B by the high affinity Sso7d
to the DNA strand. As another example, we successfully developed a novel
processive PCR method, using the archaeal Pol B with the help of a mutant PCNA.
Several DNA sequencing technologies, referred to as “next-generation
sequencing”, have been developed, and are now commercially available. molecule
detection, using dye-labeled modified nucleotides and longer read lengths, is
now known as “third-generation DNA sequencing”. These technologies apply DNA
polymerases or DNA ligases from various sources, indicating that these DNA
replication enzymes are indispensable for the development of DNA manipulation
technology. These facts prove that the progress of the basic research on the
molecular biology of archaeal DNA replication will promote the development of
the new technologies for genetic engineering.
Russian Translation
Репликация ДНК у архей, третьего домена жизни
1. Введение
Точное дублирование и передача генетической информации являются абсолютно
необходимыми для живых организмов. Молекулярный механизм репликации ДНК был
одной из центральных тем молекулярной биологии, поэтому прилагались постоянные
усилия для выяснения точного молекулярного механизма репликации ДНК, которые
проводились после открытия структуры двойной спирали ДНК в 1953 году. Белковые
факторы, которые функционируют в процессе репликации ДНК, были определены на
сегодняшний день в трех доменах жизни (рис. 1).
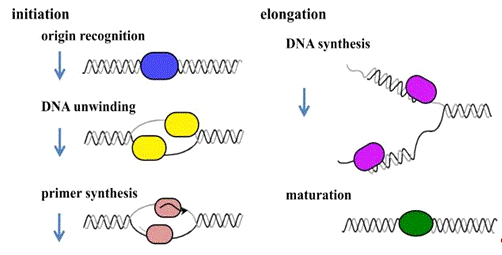
Рисунок 1. Стадии репликации ДНК
Археи, третий домен жизни, очень интересные живые организмы для изучения
из аспектов молекулярной и эволюционной биологии. Быстрый прогресс и полный
анализ генома позволили нам выполнить сравнительные исследования генома. Кроме
того, недавние экологические исследования показали, что археи обитают не только
в экстремальных условиях, но и в более обычной среде обитания. В таких
ситуациях биология архей является одной из самых захватывающих областей
исследования.
Клетки архей имеют одноклеточную ультраструктуру без ядра, напоминающую
бактериальные клетки, но белки, участвующие в генетических путях обработки
информации, в том числе в репликации, транскрипции и трансляции ДНК, во многом
схожи с теми же белками, что и у эукариот. Таким образом, большинство архейных
белков идентифицируются как гомологичные ко многим эукариотическим белкам
репликации, в том числе ORC (точка инициации транскрипции), Cdc6, GINS
(Sld5-Psf1-Psf2-Psf3), МСМ (поддержание минихромосом) RPA (репликация белка А), PCNA (ядерный антиген
пролиферирующих клеток), RFC (репликационный фактор С), FEN1 (клапан
эндонуклеазы 1), в дополнение к эукариотической праймазе, ДНК-полимеразы и
ДНК-лигазы; это явно отличает их от бактериальных белков, и эти белки
биохимически характеризуются. Их сходство показывает, что механизмы репликации
ДНК архей и эукариот произошли от общего предка, который отличался от бактерий.
Таким образом, организмы архей могут служить хорошими примерами и
разъяснить функции каждого из компонентов эукариотического типа в комплексе
механизмов репликации. Геномные и сравнительные исследования генома
архебактерий упрощаются тем, что размер генома и количество генов архебактерий
намного меньше, чем у эукариот. Механизм репликации у архей, вероятно, более
упрощен, чем у эукариот. С другой стороны, интересным фактом является то, что
кольцевая структура генома сохраняется у бактерий и архей и отличается от
линейной формы эукариотических геномов. Эти особенности побудили нас изучить
механизм репликации ДНК архей, в надежде получить фундаментальное понимание
этого молекулярного механизма и его технику с эволюционной точки зрения.
Изучение репликации бактериальной ДНК на молекулярном уровне началось
примерно в 1960 году, а затем исследования эукариот проводились с 1980 года.
Так как позже археи были признаны третьим доменом жизни, исследования
репликации ДНК архей стали активно проводиться после 1990 года. С увеличением
имеющейся общей последовательности генома, процесс исследования репликации ДНК
архей был быстрым, и глубина наших знаний о репликации ДНК архей почти
сопоставима со знаниями в бактериальных и эукариотических областях
исследований. В этой главе мы обобщим наши текущие знания о репликации ДНК у
архей.
2. Инициация репликации
Основной механизм репликации ДНК был предсказан в "теории
репликона" Якоба и др. Они предположили, что фактор инициации признает
репликатор, который сейчас называют инициатором репликации, чтобы начать
репликацию хромосомной ДНК. Затем инициатор репликации ДНК E.coli был идентифицирован как ORIC
(начало хромосомы). Инициатор репликации архей был выявлен в Pyrococcus
abyssi в 2001 году как первый инициатор начала репликации архей.
Инициатор был расположен вверх по течению от гена, кодирующего Cdc6 и
ORC1-подобные последовательности в геноме Pyrococcus. Мы обнаружили ген,
кодирующий аминокислотную последовательность, которая имела сходство с
эукариотическими Cdc6 и ORC1, которые являются эукариотическими инициаторами.
Убедившись, что этот белок фактически связывается в ORIC области на
хромосомной ДНК, мы назвали генный продукт Cdc6/Orc1, из-за его примерно равной
гомологии с областями эукариотических ORC1 и Cdc6. Ген состоит из оперона и
гена распознавания ДНК полимеразы D (она была первоначально названа Pol II, как
вторая ДНК-полимераза в Pyrococcus furiosus) в геноме.
ORIC является сохраненными повторами 13 связывающих белков, как предсказано
ранее биоинформатиками, и два из повторов больше и окружают предполагаемый DUE (элемент раскручивания ДНК) с
AT-богатых последовательностей в геномах Pyrococcus (рис. 2). Более
длинная повторяющаяся последовательность обозначается как ORB (фактор инициации
распознавания), и это было фактически доказано в исследовании Cdc6/Orc1 у Sulfolobus
solfataricus. Повтор13 связывающих белков называется miniORB, как минимальная
версия ORB. Полный анализ микрообластей генома P. abyssi показал,
что Cdc6/Orc1 связывается с ORIC областью с крайней специфичностью, и
специфическое связывание P. furiosus Cdc6/Orc1 в ORB и miniORB было подтверждено в
пробирке. Следует отметить, что многообразие точек инициации было
определено в геноме Sulfolobus. В настоящее время хорошо известно, что
есть три точки инициации у Sulfolobus, и они одновременно принимают
участие в клеточном цикле.
Анализ механизма того, как несколько точек инициации используются для
репликации генома является интересной темой в научно-исследовательской области
изучения репликации ДНК архей. Главный вопрос заключается в том, как
регулируется инициация репликации из нескольких точек инициации, и как
протекает процесс репликационной вилки после столкновения двух вилок с
противоположных направлений.

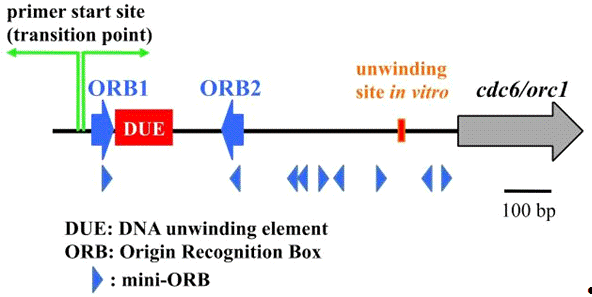
Рисунок 2. Область ORIC в геноме Pyrococcus.
3.
Как распознают Cdc6/Orc1 в ORIC?
Важным шагом в характеристике инициации репликации ДНК у архей - понять,
как белок Cdc6/Orc1 признает область ORIC. На основе разбора
аминокислотной последовательности архейных Cdc6/Orc1 установлено, что белки
принадлежат к AAA+ семье белков. Были определены кристаллическая структура
Cdc6/Orc1 белка из Pyrobaculum aerophilum и один из двух Cdc6/Orc1
белков, ORC2 из Aeropyrum Pernix (два гомолога в этом организме авторы
называют ORC1 и ORC2 ). Эти Cdc6/Orc1 белки состоят из трех структурных
доменов.
Домены I и II принимают складчатую структуру в семье AAA + белков.
Свернутая крылатая спираль (WH), которая показывает число ДНК-связывающих
белков, находится в области III. Четыре ORB расположены попарно с обеих сторон DUE в области ORIC у А. Pernix,
и ORC1 связывается с каждым ORB в виде
димера. Был предложен механизм, в котором ORC1 связывает все четыре ORB, чтобы
ввести более высокий порядок сборки для разматывания DUE, с изменениями как в топологии, так и в
суперспиральности. Кроме того, установлено, что кристаллическая структура
Cdc6-1 и Cdc6-3 у С. solfataricus (два из трех Cdc6/Orc1 белков в
этом организме) с образованием гетеродимера связана с ori2 ДНК (один из
трех истоков в этом организме), и была определена область ORC1 у А. Pernix,
связанная с последовательностью происхождения. Эти исследования показали, что
N-концевой AAA + АТФазы домен (домен I + II) и С-концевой WH домена (домен III)
способствуют процессу связывания ДНК и структурной информации не только
определением полярности инициатором сборки на происхождение, а также указывают
существенные искажения индукции, которые, вероятно, вызывают раскручивание
ДНК-дуплекса, чтобы начать процесс репликации в ДНК. Эти структурные данные
также предоставляют подробный режим взаимодействия между белком инициатора и ORIC
в ДНК.
Мутационный анализ Methanothermobactor thermautotrophicus Cdc6-1
белка показал существенное взаимодействие между остатком аргинина, который
сохраняется в архейных Cdc6/Orc1 и инвариантный гуанин в последовательности
ORB. Область Cdc6/Orc1 в P. furiosus трудно привести в
растворимую форму. Определенный сайт в ORIC, соответствующий
раскручиванию в лабораторных условиях, был определен с помощью белка,
подготовленного к процедуре денатурации-ренатурации.
Как показано на рисунке 2, область раскручивания составляет около 670 пар
оснований от области перехода между ведущими и отстающими синтезами, которые
были определены ранее в естественных условиях в точке инициации
репликации (RIP). Хотя детали репликации, которая должна быть создана в
развернутой области, полностью не поняты у архей, ожидается, что она минимально
включает MCM, GINs, праймазы, PCNA, ДНК-полимеразы, и RPA, как описано ниже. Следующие
исследования P. furiosus показали, что АТФазы Cdc6/Orc1 белка
были полностью подавлены путем связывания с ДНК, содержащей ORB.
Эксперименты на ограниченный протеолиз и ДНКаза-I-отпечаток предположили,
что Cdc6/Orc1 белок изменяет свою конформацию на последовательность ORB в
присутствии АТФ. Физиологический смысл этого конформационного изменения не
установлен, но оно должно выполнять важные функции, чтобы начать процесс
инициации, как и в случае бактериального белка DnaA. Кроме того, результаты
анализа в пробирке показали, что MCM (MCM белковый комплекс) в
геликаза-репликативной ДНК, полученной в ORIC области, является
Cdc6/Orc1-зависимым, но не АТФ-зависимым. Однако, этого сигнала недостаточно
для активации функции раскручивания MCM, и некоторые другие функции еще
предстоит определить, чтобы функциональная нагрузка этой геликазы содействовала
прогрессированию вилки репликации ДНК.
4.
MCM геликаза
После разматывания области ORIC, репликативная геликаза должна
оставаться загруженной, чтобы обеспечить непрерывное раскручивание
двухцепочечной ДНК по мере продвижения вилки репликации в двух направлениях.
Комплекс белка MCM, состоящий из шести субъединиц (Mcm2, 3, 4, 5, 6, 7), как
известно - «ядро» репликативной геликазы в эукариотических клетках. далее
взаимодействует с Cdc45 и GINS с образованием тройной сборки, называющейся
"CMG комплекс", который, как полагают, является функциональной
геликазой в эукариотических клетках (рис. 3). Однако эта идея до сих пор не
применима для эукариотических репликативных геликаз.

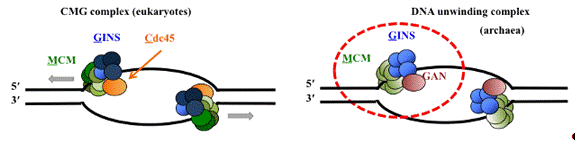
Рисунок 3. ДНК-Амортизация комплекса у эукариот и архей
Комплекс CMG является репликативной геликазой для шаблона реакции
раскручивания ДНК у эукариот. Геномы архей содержат гомологи MCM, GINS и белки, но гомологи Cdc45 не были идентифицированы.
Последние исследования показывают, что RecJ-фильная экзонуклеазная GAN, которая имеет слабую гомологическую
последовательность, что и Cdc45, может работать в качестве геликазного
комплекса с MCM и GINS.
Большинство архейных геномов, по-видимому, кодируют по меньшей мере один
Mcm гомолог и геликазу, и активность этих белков из нескольких архейных
организмов была подтверждена в пробирке. В отличие от эукариотических
MCM, архейные MCM состоят из гомогексамера или двойного гомогексамера, которые
имеют различные активности геликазы ДНК сами по себе в пробирке, и,
следовательно, эти MCM сами по себе могут функционировать как репликативная
геликаза в естественных условиях.
В настоящее время проводится большое количество исследований
Структурно-функциональных отношений в архейных MCM с использованием очищенных
белков и сайт-направленного мутагенеза. Первоначальный отчет использования ChIP метода показал, что белок MCM у P. Abyssi
преимущественно связывается с инициацией в естественных условиях в
экспоненциально растущих клетках. MCM геликаза в P. furiosus не
отображает значительной активности геликазы в пробирке. Однако
активность ДНК геликазы явно стимулировали добавлением GINS (Gins23-Gins51
комплекс), который является гомологом эукариотического GINS комплекса (описано
ниже более подробно). Этот результат позволяет предположить, что работа
комплекса с другими сопутствующими факторами в ядре комплекса в P. Furiosus
похожа на эукариотический комплекс CMG, как описано выше.
Некоторые археи имеют более двух Cdc6/Orc1 гомологов. Было обнаружено,
что два Cdc6/Orc1 гомолога, Cdc6-1 и Cdc6-2, подавляют геликазную активность
MCM в M. thermautotrophicus. Аналогично, Cdc6-1, ингибирует
активность в MCM у С. solfataricus. В противоположность этому,
Cdc6-2 белок является стимулятором геликазной активности MCM в Thermoplasma
acidophilum. Функциональное взаимодействие между Cdc6/Orc1 и белками МСМ
должно быть исследовано более подробно для достижения более глубокого понимания
сохранения и разнообразия механизмов инициирования репликации ДНК у архей.
Еще одна интересная особенность начала репликации ДНК заключается в том,
что несколько архей имеют несколько генов, кодирующих гомологи MCM в их геномах. На основе последнего
комплексного анализа генома, тринадцать видов архей имеют более одного MCM гена. Тем не менее, многие из MCM генов в геноме архей, кажется,
находятся внутри подвижных элементов, имеющих вирусное происхождение. Например,
два из трех генов в геноме Thermococcus kodakarensis располагаются в
областях, куда генетические элементы, возможно, были интегрированы.
Создание системы генетических манипуляций для Т. Kodakarensis является
первой для гипертермофильных эвриархей и выгодно для исследования функции этих MCM белков. Две группы недавно провели
эксперименты по разрушению генов для каждого гена MCM. Эти эксперименты показали, что штамм-нокаут для MCM1
и MCM2 было легко выделить, но MCM3 не может быть нарушена. Содержится значительное количество MCM 3 в клетках Т. Kodakarensis.
Кроме того, лабораторный эксперимент с использованием очищенного белка MCM показал, что только форма MCM 3 обладает стабильной гексамерной
структурой в растворе. Эти результаты подтверждают утверждение, что MCM 3 является основным белком в
основной геликазе в нормальном процессе репликации ДНК у T. kodakarensis.
Функции двух других белков MCM до
конца не изучены. Гены для MCM1 и MCM 2 стабильно передаются по наследству, и их генные продукты могут
выполнять некоторые важные функции в метаболизме ДНК у T. kodakarensis.
Активность ДНК геликазы рекомбинантного белка MCM1 обладает высокой активностью
в лабораторнцх условиях, и различные количества белка MCM1 присутствует
в клетках T. kodakarensis. Кроме того, MCM1 функционально
взаимодействует с комплексом GINS из T. Kodakarensis. Эти
наблюдения являются убедительным доказательством того, что MCM1 действительно
участвует в некоторых процессах ДНК, а так же может быть замещена MCM 3.
Наши эксперименты показали, что иммунопреципитация MCM1 сопоставима с MCM 3 и GINS, и хотя они не образуют
гетерогексамерного комплекса, предполагают, что MCM1 участвует в реплисоме или
репарсоме и выполняет некоторые функции в клетках Т. kodakarensis.
Хотя западный блот-анализ не смог обнаружить MCM 2 в выписке из экспоненциально растущей клетке T.
kodakarensis, RT-PCR эксперимент обнаружил наличие следов гена MCM2
в клетках. Рекомбинантный белок MCM 2
также имеет АТФазы и геликазы деятельности в пробирке. Таким образом,
ген MCM2 экспрессируется в нормальных условиях роста и может работать в
некоторых процессах с быстрой отдачей. В будущем необходимы эксперименты по
измерению эффективности транскрипции гена MCM2 на количественной ПЦР, а также оценка стабильности
белка MCM2 в экстракте клеток.
Фенотипический анализ исследования чувствительности Δ
MCM1 и Δ MCM2 мутантных штаммов на повреждения
ДНК, вызванными различными мутагенами, как сообщалось для других репараций ДНК
генов, связанных в Т.kodakarensis, может дать ключ к выяснению функции
белков MCM. Methanococcus maripaludis S2
имеет области четырех MCM в своем геноме, три из которых, кажется, быть
получены из фага, направленное протеомическое исследования выявило пептиды,
происходящих из трех из четырех продуктов гена MCM. Кроме того, четыре генных продукта совместной
экспрессии в клетке E.coli совместно очищаются таким же фракциями. Эти результаты позволяют
предположить, что несколько белков MCM являются функциональными в клетках М. maripaludis.
5.
Комплектование MCM в области ORIC
Другой важный вопрос - как MCM комплектуется на развернутой области ORIC.
Детальный механизм загрузки геликазы MCM не выяснен. Считается, что
архебактерии используют различные механизмы сборки геликазы MCM в ORIC.
Анализ набора в лабораторных условиях показал, что MCM у P. Furiosus комплектует ORIC ДНК
Cdc6/Orc1-зависимым образом. Этот анализ показал, что предварительная загрузка
Cdc6/Orc1 на ORB ДНК приводит к явному снижению комплектации MCM в регионе
ORIC, полагая, что свободные Cdc6/Orc1 предпочтительнее в качестве геликазного
сборщика, сообщающегося с MCM и доводящий его до ORIC. Было бы интересно
понять, каким образом две задачи, инициация распознавания и комплектование MCM,
выполняются в белке Cdc6/Orc1, так как домен WH, который в первую очередь
распознает и связывает ORB, также имеет сильное сродство к белку MCM.
Сборка белка MCM на ДНК ORB Уокер-мутанта P. furiosus
в Cdc6/Orc1 произошла с той же эффективностью, что и в немутантных типах
Cdc6/Orc1. В случае ДНК-связывания Cdc6/Orc1 у P. Furiosus имеет сходство в присутствии и в отсутствии
АТФ, как и в случае инициации белков из Archaeoglobus fulgidus, С.
solfataricus, А. Pernix. Так что до сих пор не известно,
является ли обязательным гидролиз АТФ, и регулирует ли активность Cdc6/Orc1
набор белков MCM на ORIC в клетках.
Еще одной важной проблемой является очень низкая эффективность сборки
белков MCM, о чем стало известно в лабораторных
исследованиях. Количественная оценка сборки белка MCM в лабораторных условиях показала, что в ORB было собрано менее одного гексамера MCM. Стало известно, что линейные ДНК,
содержащие ORB1 и ORB2, используемые в проверке сборки, не могут быть пригодны
для восстановления механизма репликации ДНК у архей, и может потребоваться
шаблон, который будет более точно имитировать хромосомную ДНК.
Кроме того, может оказаться, что еще не идентифицированы белки,
необходимые для достижения эффективной сборки геликазы в клетках P. furiosus
в лабораторных условиях. Наконец будет необходимо построить в лабораторных
условиях более определенную систему репликации для анализа регуляторных функций
Cdc6/Orc1 именно в процессе инициации репликации.
В М. thermautotrophicus белки Cdc6-2 могут отделяться мультимерами
MCM. Cdc6-2 может потребоваться в
качестве грузчика MCM геликазы в организме. Взаимодействие между Cdc6/Orc1 и MCM, вероятно, общее для изучаемых
организмов. Тем не менее, влияние Cdc6/Orc1 на активность геликазы MCM
отличается у различных организмов, как описано выше. Некоторые другие белковые
факторы могут действовать у различных архей, например белок, отдаленно связаный
с эукариотической Cdt1, который играет решающую роль во время комплектации MCM
у эукариот, существует у некоторых архей, хотя его функции еще не были
охарактеризованы.
6.
GINS
Эукариотический комплекс GINS был первоначально идентифицирован в Saccharomyces
cerevisiae как существенный белковый фактор для
инициации репликации ДНК. GINS состоит из четырех различных белков, Sld5, Psf1,
Psf2 и Psf3 (поэтому, GINS
является акронимом от японского go-ichi-ni-san, то есть цифры
5-1-2-3 после этих четырех субъединиц). Сохранение последовательности
аминокислот из четырех субъединиц в комплексе GINS предполагает, что они
являются паралогами предковой формы. Тем не менее, большинство архейных геномов
имеют только один ген, кодирующий этот белок, и, что более интересно,
Crenarchaeota и Euryarchaeota (два основных рода архей) имеют два гена с
последовательностями, аналогичными Psf2 и Psf3 и Sld5 и Psf1, их соответственно
называют GINS23 и GINS51.
Гомолог GINS, обозначенные как GINS23, был обнаружен биохимически в С.
solfataricus как первый белок GINS в археях, в процессе дрожжевого двухгибридного скрининга взаимодействия
партнеров MCM белка и другой субъединицы,
обозначенной как GINS15, был
идентифицирован масс-спектрометрическим анализом иммуноаффинно-очищенного
нативного GINS из экстракта S. Solfataricus. У S. solfataricus GINS состоит из двух белков,
Gins23 и Gins15, и образует тетрамерную структуру с молярным соотношением 2:2.
GINS из P. furiosus, комплекс GINS23 и GINS51,
с соотношением 2:2, был определен в качестве первого эвриархейного комплекса
GINS. Было отдано предпочтение белку GINS51, потому что GINS15
не соответствовал порядку цифр в аббревиатуре GINS.
Гексамер MCM2-7 был сопоставлен в комплексе с Cdc45 и GINS из зародышей Drosophila melanogaster и S. cerevisiae, а "CMG (Cdc45-MCM2-7-GINS)
комплекс", как описано выше, должен быть важен для функции репликативной
геликазы. Комплекс CMG был также связан с репликативной вилкой в экстрактах яиц
Xenopus laevis, и большой молекулярный механизм,
содержащий Cdc45, GINS и MCM2-7, был предложен в качестве анвиндосомного
отделителя ДНК в репликативной вилке.
Поэтому, GINS должны быть
решающим фактором не только для процесса инициации, но и процесса пролонгации
эукариотической репликации ДНК. Так как комплекс GINS у S. solfataricus
взаимодействует с MCM и праймазой, предполагается, что GINS состоит в
реплисоме. Конкретные функции GINS в реплисоме еще предстоит определить.
Стимуляции или ингибирования геликазной или примазной активностей при
взаимодействии GINS S. Solfataricus в лабораторных условиях не
наблюдалось. С другой стороны, активность MCM геликазы ДНК P. Furiosus явно стимулировали добавлением к P.
Furiosus комплекса GINS,
как описано выше.
В отличие от S. solfataricus
и P. furiosus, каждый из которых экспрессирует GINS23 и GINS51, Thermoplasma acidophilum имеет один гомолог
GINS, GINS51. Рекомбинантный белок из GINS51 T. Acidophilum принял форму
гомотетрамера, что было определено при помощи гель-фильтрации и электронной
микроскопии. Кроме того, физическое взаимодействие между T. acidophilum
GINS51 и MCM было обнаружено при помощи поверхностного плазмонного
резонансного анализа (SPR). Несмотря на то, что GINS51 у T. acidophilum не влияет на
геликазную активность, его родственные MCM, при равном соотношении каждой
молекулы, были испытаны в лабораторных условиях, и избыточное количество Gins51
ясно стимулировало геликазную активность. В случае T. kodakarensis,
АТФазы и геликазная активность MCM1 и MCM3 явно стимулируется GINS T. Kodakarensis в лабораторных условиях.
Интересно, что деятельность геликазы MCM1 имеет более сильный стимул, чем у
MCM3.
Также были обнаружены физические взаимодействия между GINS T. Kodakarensis и белками МСМ. Эти сообщило о
том, что МСМ-GINS комплекс является общей частью репликативной геликазы у
архей. Недавно определенная кристаллическая структура тетрамера GINS в T. Kodakarensis,
состояая из GINS51 и GINS23, была определена как схожая со структурой GINS человека. Каждая субъединица GINS
человека разделяет аналогичные фрагменты, и собирается в гетеротетрамер
уникальной трапециевидной формы. Sld5 и Psf1 обладают α-спиральным (A) доменом на N-конце и β-многожильным доменом (B) на С-конце
(AB-тип).
С другой стороны, Psf2 и Psf3 являются перестановкой предыдущей версии
(BA-тип). Основные структуры каждой субъединицы и тетрамерной сборки GINS T.
kodakarensis аналогичны GINS человека. Однако, расположение С-концевого
домена B из GINS51 удивительно различается между
двумя структурами GINS.
Гомологичная модель гомотетрамерного GINS из T. acidophilum
была собрана с использованием кристаллической структуры GINS T. kodakarensis
в качестве шаблона. GINS51
имеет длинную неупорядоченную область, вставленную между А и В областями, и это
позволяет конформации С-концевых доменов быть более гибкой. Это расположение
домена приводит к образованию асимметричного гомотетрамера, вместо симметричной
собрки GINS в T. kodakarensis.
белок повсеместно распространен от дрожжей до человека, что подтверждает
мнение, что образование комплекса CMG является универсальным в эукариотических
процессах репликации ДНК. Однако никаких гомологов Cdc45 у архей выявлено не
было. Недавний доклад о биоинформатическом анализе показал, что первичная
структура эукариотической и прокариотической Cdc45 RecJ имеют общих предков.
Действительно, гомолог ДНК-связывающего домена RecJ был совместно очищен с
помощью GINS из S. solfataricus.
Наш опыт обнаружил стимуляцию 5'-3' экзонуклеазной активности гомологов
RecJ из P. Furiosus и Т. Kodakarensis по
родственным комплексам GINS. Гомолог RecJ из T. kodakarensis
образует стабильный комплекс с GINS,
а экзонуклеазная деятельность 5'-3' усиливается в лабораторных условиях,
поэтому гомолог RecJ в недавней работе был обозначен как GAN, от GINS-Associated Nuclease.
Другой доклад отмечает, что структура Cdc45 человека, полученная анализом
малого угла рассеивания рентгеновских лучей (МУР) согласуется с
кристаллографической структурой семьи RecJ. Эти результаты будут способствовать
дальнейшему исследованию структуры и функции высшего порядка анвиндосом в
археях и в эукариотических клетках.
7.
Праймазы
В инициации синтеза цепи ДНК праймаза необходима для синтеза короткого
олигонуклеотида в качестве праймера. DNAG и белки p48-p58 являются праймазами у
бактерий и эукариот соответственно. P48-p58 праймаза далее образует комплекс с
p180 и p70, чтобы сформировать ДНК-полимеразу α-праймазного комплекса. Каталитические
субъединицы эукариотической (p48) и архейной праймаз отчасти схожи, но имеют
отчетливую гомологичную последовательность с такими же праймазами семейства
X-полимеразы ДНК.
Впервые архейная праймаза была идентифицирована с Methanococcus
jannaschii, как ORF с последовательностью такой же, как у эукариотической
p48. Продукты гена проявили активность ДНК-полимеразы и смогли синтезировать
олигонуклеотиды на матричной ДНК.
Мы характеризовали p48-подобный белок (p41) из P. furiosus.
Неожиданно было установлено, что у архей белок p41 не синтезирует короткие РНК
сам по себе, но преимущественно использует дезоксинуклеотиды, позволяющие
синтезировать ДНК до несколько тысяч пар нуклеотидов в длину. Кроме того, ген,
находящийся по соседству с геном p41, кодирует белок с очень слабым сходством с
p58-субъединицей эукариотической праймазы. Генный продукт, обозначенный p46, в
лабораторных условиях фактически образует стабильный комплекс с p41, и комплекс
может синтезировать короткие РНК праймеры, а также ДНК из нескольких сотен
нуклеотидов.
Короткие РНК, но не ДНК-праймеры, были обнаружены в клетках Pyrococcus,
так что можно предположить, что некоторые механизмы преимущественно
использовали РНК-праймеры, которые существуют в клетках.
Дальнейшие исследования по гомологам праймаз из S. solfataricus,
Pyrococcus horikoshii, P. Abyssi,
в лабораторных условиях дали схожие результаты. Примечательно, что p41
является каталитической субъединицей, и он один модулирует активность в
гетеродимерных праймазах архей.
Малые и большие субъединицы также называют PRIS и PRIL соответственно. Кристаллическая структура
N-концевого домена PRIL, объединенной с PRIL, в праймазе S. solfataricus показала, что PRIL не
контактирует непосредственно с активным сайтом PRIS и, следовательно, большие
субъединицы могут взаимодействовать с синтезированными праймерами, для
регулировки его длины до 7-14 мер. Структура каталитического центра аналогична
полимеразе семейства X-ДНК.
'-концевая нуклеотидная трансферазная активность, обнаруженная в праймазе
S. solfataricus, а также заполнение пробелов и странд-смещение
деятельности в праймазе P. abyssi также поддерживают структурное
сходство между PRIS и семейством
X-полимераз ДНК. Уникальная область, названная PADT, обнаруженная в комплексе PRISL в S.solfataricus,
была научно опубликована совсем недавно.
Область может быть включена в восстановление двунитевой цепочки у архей.
Архейный геном также кодирует последовательность, похожую на бактериальную
праймазу типа DNAG. Гомолог DNAG из генома P. furiosus была
выделен в E. coli, но в
лабораторных условиях белок не показал активность синтеза праймера, и таким
образом, архейный DNAG-подобный белок не может действовать в качестве праймазы
в клетках Pyrococcus.
Исследованием DNAG-подобного белка было доказано участие в деградации
РНК, как компонента экзосомы. Тем не менее, в недавно опубликованной статье
сообщается, что гомолог DNAG из S. solfataricus фактически синтезирует
праймеры с 13 нуклеотидных цепочек. Было бы интересно узнать, если две
различные праймазы разделят праймер синтеза для ведущих и отстающих нитей
репликации, в клетке Sulfolobus, по предположению авторов.
Интересна предлагаемая гипотеза об эволюции PriSL и DNAG из последнего
универсального общего предка (LUCA).
Белок PriSL в клетке Sulfolobus продемонстрировал как происходит
взаимодействие с MCM через GINS23. Это праймазно-геликазное
взаимодействие, вероятно, обеспечивает связь раскручивания ДНК и праймера при
продвижении вилки репликации.
Кроме того, непосредственное взаимодействие между PriSL и загрузчика RFC
(описанное ниже) в S. solfataricus может регулировать синтез
праймера и передачи его в ДНК-полимеразные клетки архей.
8.
Одноцепочечный ДНК-связывающий белок
Одноцепочечный ДНК-связывающий белок, который называется SSB у бактерий и
архей и RPA у эукариот, является важным фактором
для защиты разматывания одноцепочечной ДНК при нуклеазной атаке, химических
модификаций и других нарушений во время процессов репликации и репарации ДНК.
SSB и RPA имеют структурно подобные домены,
содержащие общие фрагменты, называемые OB (олигонуклеотид /
олигосахарид-связывающие) фрагменты, хотя и имеют малое сходство в своих
аминокислотных последовательностях.
Общая структура позволяет предположить, что механизм одноцепочечного
ДНК-связывания сохраняется в живых организмах, несмотря на отсутствие сходства
последовательностей. Белок SSB у E. coli является
гомотетрамером от 20 кДа пептида с одним ОВ-фрагментом и SSB от Deinococcus
radiodurans и Thermus aquaticus, состоящий из гомодимера пептида, содержащего два
OB-фрагмента.
Эукариотическая RPA является стабильным гетеротримером, состоящим из 70,
32 и 14 кДа белка. RPA70 содержит два тандемных повтора ОВ-фрагмента, которые
отвечают за основные взаимодействия с одноцепочечной ДНК в ее центральной
области. N-концевые и С-концевые области RPA70 осуществляют взаимодействие с
RPA32, а также со многими клеточными или вирусными белками. RPA32 содержит
ОВ-фрагменты в центральной и С-концевой областях взаимодействия с другими
субъединицами РПА и различными клеточными белками. RPA14 также содержит
ОВ-фрагменты.
Эукариотические RPA взаимодействуют с SV40 Т-антигеном и ДНК-полимеразами
α-примазного комплекса, и таким образом
формируют часть комплекса инициации при репликации. RPA также стимулирует Polα-праймазную активность и
PCNA-зависимые Polδ активности. RPA от М. jannaschii и М. thermautotrophicus
были зарегистрированы в 1998 году, как первый одноцепочечный ДНК-связывающий
белок архей. Эти белки имеют сходства их аминокислотных последовательностей с
эукариотической RPA70 и содержат четыре или пять повторов ОВ-фрагмента и один
цинк-направляющий фрагмент.
RPA в М.jannaschii
существует в виде мономера в растворе и имеет однонитевой ДНК-связывающий
фрагмент. С другой стороны, RPA P. furiosus
образует комплекс, состоящий из трех отдельных подразделений, RPA41, RPA32 и
RPA14, похожих на эукариотические RPA. В лабораторных условиях RPA в P. furiosus
поразительно стимулирует RADA-раскрученные
обменные реакции.
В то время как у эвриархей имеются гомологи эукариотического типа RPA, кренархейные белки SSB оказываются
гораздо более связанными с бактериальными белками, с одной стороны имея
OB-фрагмент и с другой стороны С-концевой хвост. Однако кристаллическая
структура белка SSB из S. solfataricus показала, что ОВ-фрагменты
домена больше похожи на эукариотические гомологи RPA, что поддерживает тесную связь между археями и
эукариотами.
RPA из
Methanosarcina acetivorans демонстрирует уникальные свойства. В отличие
от нескольких белков RPA в
других архебактериях и эукариотах, каждая субъединица RPA в M.acetivorans - RPA1, RPA2 и RPA3 являются
4, 2 и 2 OB-фрагментами соответственно, и могут выступать в качестве отдельных
одноцепочечных ДНК-связывающих белков. Кроме того, каждый из трех белков RPA, а также их комбинации, очевидно,
стимулируют активность удлинения праймера BI ДНК-полимеразы М. acetivorans,
как было показано ранее в лабораторных условиях для бактериальных SSB и эукариотических RPA.
Архитектура SSB и RPA
предполагает, что они состоят из различных комбинаций OB-фрагментов. Бактериальные и эукариотические организмы
содержат один тип SSB или RPA
соответственно. С другой стороны, организмы архей имеют различные RPA, состоящие из различных организаций
ОВ-фрагментов. Гипотезу, что гомологичная рекомбинация может играть важную роль
в создании такого разнообразия OB-фрагментов в клетках архей была предложена на
основе экспериментов, характеризующих соединение RPA с различными OB-фрагментами.
9.
ДНК-полимераза
ДНК-полимераза катализирует образование фосфодиэфирной связи между
терминалом 3'-ОН праймазы и α-фосфатом входящего трифосфата для
продления короткого праймера, и, следовательно, основного игрока в процессе
репликации ДНК. На основе сходства их аминокислотных последовательностей
ДНК-полимеразы были разделены на семь семей: A, B, C, D, E, X и Y.
Фундаментальная способность ДНК-полимеразы синтезировать
дезоксирибонуклеотидные цепи в целом сохраняется, но более конкретные свойства,
в том числе процессивность, точный синтез и подложка нуклеотидной селективности
различаются в зависимости от семьи. Ферменты в пределах одной семьи имеют в
основном схожие свойства. E. coli имеет пять ДНК-полимераз, Pol I, Pol
II и Pol III, которые относятся к семьям A, В и С соответственно. Pol IV и Pol V классифицируются в семье Y, в качестве ДНК-полимеразы для
трансповреждающего синтеза (TLS). У эукариот имеются репликативные
ДНК-полимеразы, Pol α, Pol δ и Pol ε, принадлежащие семье B, и
трансповреждающие ДНК-полимеразы, η, ι и κ, принадлежащие семье Y.
Наиболее интересную особенностью, обнаруженной к моменту начала этой
области исследований было то, что археи действительно имеют эукариотические Pol
α-подобные (Семья B) ДНК-полимеразы.
Члены кренархеот имеют как минимум две ДНК-полимеразы семьи B. С другой стороны, существует только
одно семейство B ДНК-полимераз у эвриархей. Вместо этого геномы эвриархей
кодируют ДНК-полимеразы семьи D, предлагаемые в качестве Pol D, которая кажется
специфичной для этих архей, и никогда не была найдена в других областях.
Гены семейства Y-подобных ДНК-полимераз сохраняются в нескольких, но не
всех, архейных геномах. Роль каждой ДНК-полимеразы в клетке архей сих пор не
известна, однако ДНК-полимеразы получают четкое распределение.
Первое семейство D ДНК-полимераз было определено из P. furiosus,
путем скрининга ДНК-полимеразной активности в клеточном экстракте.
Соответствующий ген был клонирован, что продемонстрировало, что эта новая
ДНК-полимераза состоит из двух белков, названных DP1 и DP2, и выведенные
аминокислотные последовательности этих белков не сохраняются в ДНК-полимеразах
их семей. Pol D в P. furiosus демонстрирует эффективную
деятельность в развертывании нитей и сильной корректурой дальнейшей
деятельности.
Другое семейство D-полимераз в ДНК было также охарактеризовано
несколькими группами. Pol D гены были найдены только в эвриархеотах. Однако
последние данные экологической геномики и увеличение усилий открыли новые виды
у архей: таумархеоты, корархеоты и аигархеоты, и были открыты их
последовательности генома, имеющие гены, кодирующие Pol D. Генетическое
исследование на Halobacterium Sp. NRC-1 показали, что и Pol B, и Pol D - обе они имеют важное значение для
жизнеспособности.
Интересным вопросом является выяснение того, что Pol B и Pol D работают вместе на репликационной
вилке для синтеза ведущих и отстающих нитей соответственно. В соответствии с
использованием РНК-праймера и наличием перемещения области активности, Pol D
может катализировать синтез отстающей нити.
Таумархеоты и аигархеоты имеют гены, кодирующие Pol D и кренархейную Pol
ВII, в то время корархеота кодирует Pol
BI, Pol BII и Pol D.
Биохимическая характеристика этих генных продуктов будет способствовать
исследованиям эволюции ДНК-полимеразы в живых организмах.
Была предложена гипотеза о том, что археи являются предком эукариот,
кодирующих три ДНК-полимеразы из двух различных семьи полимераз:
B-ДНК-полимеразу и D-ДНК-полимеразу,
и все это способствовало эволюции техники репликации эукариот, состоящей из Pol
α,
δ, и ε. Белок, кодирующийся в плазмиду PRN 1
был выделен из штамма Sulfolobus. Этот белок, ORF904 (названный RepA),
осуществляет свою деятельность праймазы и ДНК-полимеразы в N-концевом домене и
геликазную деятельность в С-концевом домене, и, вероятно, будет необходим для
репликации PRN1.
Для аминокислотной последовательности N-концевого домена не найдены
гомологи с любой известной ДНК-полимеразой или праймазой и, следовательно, было
предложено семейство E. Похожие белки кодируются различными архейными и
бактериальными плазмидами, а также некоторыми бактериальными вирусами.
Недавно один белок, tn2-12p, закодированный в плазмиде pTN2, полученный
от Thermococcus nautilus,
был экспериментально идентифицирован как ДНК-полимераза из этого семейства. Этот
фермент, вероятно, ответственен за репликацию плазмиды. Дальнейшее исследование
этого семейства ДНК-полимераз будет интересно с эволюционной точки зрения.
10.
PCNA и RFC
Скользящий механизм зажима с кольцевой структурой в форме пончика
сохраняется в живых организмах и функционирует как платформа или леса для
белков, работающих на нити ДНК. У эукариот и архей PCNA имеют гомотримерную
кольцевую структуру, которая окружает цепь ДНК и является якорем для многих
важных белков, участвующих в репликации и репарации ДНК (рис. 4). работает как
фактор процессивности, который сохраняет ДНК-полимеразы на ДНК путем связывания
их на одной поверхности (лицевой стороне) кольца для непрерывного синтеза цепи
ДНК в репликации ДНК (рис. 5). Для введения нити ДНК в центральное отверстие
зажимного кольца зажимающей структуре требуется открыть кольцо для
взаимодействия с зажимом. У архей и эукариот структура зажима называется RFC
(рис. 5).
На сегодняшний день наиболее изученными молекулами PCNA и RFC у архей
являются молекулы PCNA и RFC в P. Furiosus. Молекулы PCNA и RFC
необходимы для ДНК-полимеразы для выполнения поступательного синтеза ДНК.
Молекулярный механизм процесса загрузки зажима активно исследовался (рис. 5).
Промежуточный PCNA-RFC-ДНК, в которой кольцо PCNA открыто во
внеплоскостном режиме, было обнаружено анализом частиц
электронно-микроскопических изображений белков P. furiosus (рис.
6). Недавно были опубликованы кристаллическая структура комплекса, в том числе
АТФ-связанная структура зажима, раскрытие цикла зажима матричного праймера ДНК
с использованием белков бактериофага Т4, и знания наши о механизме запуска
зажима постоянно прогрессируют.
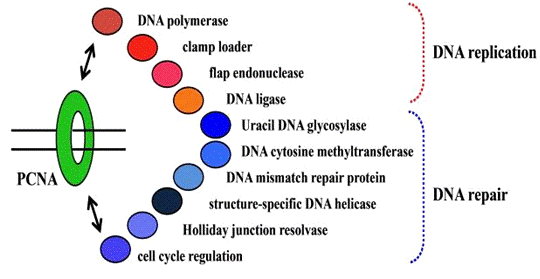
Рисунок 4. PCNA-взаимодействующие
белки.
После загрузки зажима ДНК-полимераза получает доступ к зажимам и
зажим-полимеразного комплексу, выполняющему процесс поступательного синтеза
ДНК. Таким образом, структурный и функциональный анализ ДНК-полимеразы
PCNA-комплекса является следующей целью выяснения общих механизмов репликации
прогрессивной вилкой. Взаимодействующие белки PCNA содержат небольшое
количество сжатой информации о фрагментах последовательности, называемой PIP-бокс,
который связывается с общими областями на PCNA. бокс состоит из
последовательности "Qxxhxxaa", где "х" обозначает любую
аминокислоту, "h"
представляет собой гидрофобный остаток (например, L, I или M) и "а"
обозначает ароматический остаток (например, F, Y или W). ДНК-полимеразы архей
имеют PIP-бокс-фрагменты в их последовательности.
Тем не менее, только в нескольких исследованиях экспериментально
исследовалась функция фрагментов. Была определена кристаллическая структура Pol
B в P. furiosus в виде комплекса с мономерным мутировавшим PCNA,
и была построена убедительная модель полимеразного кольца PCNA. Это
исследование показало, что новое взаимодействие образуется между растянутой
петлей PCNA и выступом области Pol B, в дополнение к аутентичному PIP-боксу.
Сравнение модели структуры с ранее изученной структурой ДНК полимеразы B-семейства из фага RB69, образующей
комплекс с ДНК предположило, что второе место во взаимодействии играет решающую
роль в переключении между полимеразным и экзонуклеазным режимами, индуцируя
составную конфигурацию PCNA-полимеразы, которая способствует синтезу после
редактирования.
Этот предполагаемый механизм для контроля точности репликативной
ДНК-полимеразы подтверждается опытом, в котором мутации на второй области
взаимодействия усиливаются экзонуклеазной активностью в присутствии PCNA. Кроме
того, трехмерную структуру тройного комплекса ДНК-полимеразы-PCNA анализировали
с помощью анализа частиц на электронном микроскопе (ЭМ).
Это структурное представление показало полную конфигурацию домена
тримерного кольца PCNA-полимеразы и ДНК, в том числе контактов типа белок-белок
или белок-ДНК. Такая архитектура обеспечивает более четкое представление в
механизме переключения между режимами редактирования и синтеза.


Рисунок 5. Механизм поступательного синтеза ДНК.
В отличие от большинства эвриархейных организмов, которые имеют один
гомотримерный гомолог PCNA, образующий циклическую структуру, большинство
кренархей имеют несколько гомологов PCNA, и они способны образовывать
гетеротримерные кольца для их функции. Особенно интересно наличие трех PCNA -
PCNA1, PCNA2 и PCNA3, специфически связывающихся с PCNA-связывающими белками, в
том числе ДНК-полимеразами, ДНК-лигазами и эндонуклеазой FEN-1.
Подробные исследования структуры гетерологичного PCNA из S.solfataricus
показали, что режимы взаимодействия между подразделениями сохраняются с
гомотримерными PCNAs. Т. kodakarensis является единственным видом
эвриархейных, который имеет два гена, кодирующих гомологи PCNA в геноме. Были
характеризованы два гена из генома T. kodakarensis, и высоко
очищенных генных продуктов, PCNA1 и PCNA2.
В лабораторных условиях PCNA1 стимулировал синтез ДНК реакции из двух
ДНК-полимераз, Pol B и Pol D из T.
Kodakarensis. Однако PCNA2 только оказал влияние на Pol B. Был выделен штамм Т. kodakarensis с
нарушениями в pcna2, в то время как нарушение гена для pcna1
добиться не удалось. Эти результаты показали, что PCNA1 необходима для
репликации ДНК, и PCNA2 может играть разную роль в клетках Т.kodakarensis.
Чувствительность Δ pcna2-мутантного штамма к ультрафиолетовому
облучению (УФ), метил-метансульфонату (MMS) и митомицину C (MMC) была
неотличима для тех же организмов из штамма дикого типа. Обе формы PCNA1 и PCNA2
имеют стабильную структуру кольца и работают в качестве фактором процессивности
в лабораторных условиях в Pol B в Т. kodakarensis.
Кристаллические структуры двух PCNA имеют различные взаимодействия на
интерфейсах субъединица-субъединица.
 С другой
стороны, архейный RFC состоит из двух белков, RFCS (маленький) и RFCL
(большой), в соотношении 4 к 1. Другая форма RFC, состоящий из трех субъединиц,
RFCS1, RFCS2 и RFCL, в соотношении 3 к 1 к 1, также была определена в M.
acetivorans. Три субъединицы RFC могут представлять собой промежуточный
этап в эволюции более сложных RFC эукариот от менее сложных RFC архей.
С другой
стороны, архейный RFC состоит из двух белков, RFCS (маленький) и RFCL
(большой), в соотношении 4 к 1. Другая форма RFC, состоящий из трех субъединиц,
RFCS1, RFCS2 и RFCL, в соотношении 3 к 1 к 1, также была определена в M.
acetivorans. Три субъединицы RFC могут представлять собой промежуточный
этап в эволюции более сложных RFC эукариот от менее сложных RFC архей.
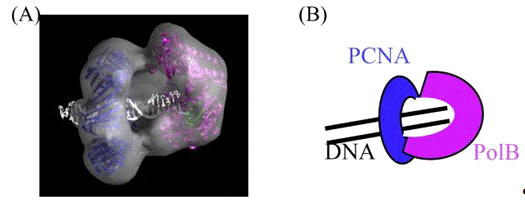
Рисунок 6. Электронно-микроскопический анализ П. furiosus
ДНК-полимераза комплекса PCNA-ДНК P. furious.
Проведен анализ и сравнение организации субъединиц и пространственного
распределения субъединиц в RFC комплексе M. acetivorans с
подобным γ-комплексом E. coli, который также пентамерен, и состоит
из трех различных белков. Эти две структуры зажима принимают аналогичные
организации субъединиц и пространственное распределение, но функции отдельных
субъединиц могут быть разнообразными.
11.
ДНК-лигазы
ДНК-лигаза подключается к фрагментам Оказаки при прерывистом синтезе
нитей при репликации ДНК, и, следовательно, универсально присутствует во всех
живых организмах. Этот фермент катализирует образование фосфодиэфирных связей с
помощью трех шагов передачи нуклеотидов. На первом этапе, ДНК-лигазы образует
ковалентную фермент-AMP промежуточную связь путем реакции с АТФ или НАД + в
качестве кофактора. На втором этапе, ДНК-лигаза признает субстратную ДНК, а AMP
затем передается от лигазы к 5'-фосфатному-концу ДНК с образованием
ДНК-аденилатного промежуточного соединения (AppDNA). На заключительном этапе,
5'-AppDNA подвергается нападению соседней 3'-гидрокси группы ДНК и образуется
фосфодиэфирная связь. ДНК-лигазы сгруппированы в две семьи, в соответствии с их
требованиями - АТФ или НАД + выступает в качестве кофактора нуклеотидов в
первой стадии реакции. ATФ-зависимые лигазы ДНК широко распространены во всех
трех областях жизни, в то время как NAD +-зависимые ДНК-лигазы существуют в
основном в бактериях. У некоторые галофильных архебактерий и эукариотических
вирусов также имеется ряд НАД +-зависимых ферментов.
На сегодняшний день в геноме человека было выделено три гена (LIG1,
LIG3 и LIG4), кодирующие
АТФ-зависимые ДНК-лигазы и ДНК-лигазы I (Lig I), кодируемых LIG1, являющиеся репликативным
ферментом, который соединяет фрагменты Оказаки при репликации ДНК. Первый ген,
кодирующий эукариотическую АТФ-зависимую ДНК-лигазу был найден в термофильных
археях, Desulfolobus ambivalens. Последующие идентификации ДНК-лигазы от
архей организмов показали, что эти ферменты в качестве кофактора в основном
используют АТФ. Тем не менее, эта классификация может быть не столь строгой.
Использование НАД +, а также АТФ, в качестве кофактора наблюдалась в нескольких
ДНК-лигазах, в том числе из T. kodakarensis, Т. fumicolans,
Р. abyssi, Thermococcus Sp. NA1, Т. acidophilum,
Picrophilus torridus и Ferroplasma acidophilum, хотя АТФ, очевидно,
предпочтительнее во всех случаях.
Двойной кофактор специфичности (ATP / NAD +) является интересной
особенностью этих ферментов ДНК-лигазы, и будет важно исследовать структурные
основы для этого. Другой двойной кофактор специфичности существует в архейных
ДНК-лигазах, которые используют ADP, а также АТФ, как обнаружено в ферментах из
А. Pernix и Staphylothermus marinus, и в случае Sulfobococcus
zilligii, ГТФ также является функциональным кофактором. ДНК-лигазы из P.
horikoshii и P. furiosus
имеют строгое предпочтение АТФ.
Достаточные биохимические данные не решили вопрос о специфике двойного
кофактора, и дальнейшие биохимические и структурные анализы обязательны для проведения.
Кристаллическая структура P. furiosus ДНК-лигазы была определена,
и было показано физическое и функциональное взаимодействие между ДНК-лигазой и
PCNA. Подробный режим взаимодействия человека Lig I и PCNA несколько
неясен, из-за нескольких спорный отчетов.
Стимулирующий эффект PCNA из P. Furiosus на ферментативную
активность родственной ДНК-лигазы наблюдался при высокой концентрации соли, в
которых ДНК-лигазы сами по себе не могут связываться с разорванным ДНК из
субстрата. Интересно, что PCNA-связывающая область расположена в середине
N-концевого ДНК-связывающего домен (DBD) из ДНК лигазы P. furiosus,
и связывающий последовательностью QKSFF, которая предлагается в качестве
короткой версии PIP-бокса на самом деле связана с поверхностью белка. Интересно,
что эта область расположена в середине цепи белка, вместо N-или С-концевой
области, где обычно расположены PIP-боксы.
Чтобы подтвердить, что эта область сохраняется в архейной / эукариотической
ДНК-лигазе, были проанализированы физические и функциональные взаимодействия
между ДНК-лигазой и PCNA в А. Pernix, и взаимодействие показало
зависимость от фенилаланина 132 остатка, который расположен в предсказанной
области от множественного выравнивания из АТФ-зависимой ДНК-лигазы.
Кристаллическая структура человеческого Lig I, в комплекс с ДНК, была
определена в качестве первой ATФ-зависимой ДНК-лигазы человека, хотя лигаза
имела усеченную N-концевую форму. Структура состоит из N-концевого
ДНК-связывающего домена, среднего домена полиаденилирования, и С-концевого
домена с ОВ-укладкой. Кристаллическая структура Lig I (остатки с 233 по 919) в
комплексе с обрезанной промежуточной 5'-аденильной ДНК показала, что фермент
перенаправляет путь дцДНК, чтобы выставить помеченные концы на реакцию
присоединения. N-концевой ДНК-связывающий домен работает, окружая субстрат ДНК
вроде PCNA и стабилизирует ДНК в искаженную структуру, позиционирующую
каталитическое ядро на метку.
Кристаллическая структура полной длины ДНК-лигазы из P. Furiosus
показала, что архитектура каждого домена напоминает их в Lig I, но домен меры
разительно отличается между двумя ферментами. Этот домен перестройки, вероятно,
связан с ролью спирального расщирения в "доменном подключении",
сохраняемом в ДНК-лигазах на С-концах в археях и эукариотах. ДНК субстрат в
открытой форме Lig I заменяет последовательность VI на С-конце, в ДНК-лигазе
закрытой формы P. Furiosus.
И форма, и электростатическое распределение подобны последовательности VI
и субстрату ДНК, что предполагает, что последовательность VI в закрытом
состоянии имитирует входящий субстрат ДНК. В последствии определена
кристаллическая структура ДНК-лигазы С. Solfataricus, которая
является полностью открытой структурой, в которой расширены три области. Также
в этой работе был проанализирован лигазный PCNA комплекс С. Solfataricus
при помощи SAXS. ДНК-лигаза С. Solfataricus связана с кольцом
PCNA, по-прежнему сохраняющим открытую, вытянутую конформацию. Закрытые,
кольцеобразные конформации наблюдается в структуре Lig I, как описано выше, что, вероятно, является активной
формой, выступающей в качестве катализатора конечных реакции соединения ДНК и,
следовательно, предполагается, что открывающее-закрывающее движение происходит
для лигирования и переключения в конформационном изменение на пластичном интерфейсе
PCNA, который служит эффективной платформой для лигирования ДНК.
В результате электронного микроскопирования был обнаружен тройной
комплекс трехмерной структуры, состоящий из ДНК-лигазы-PCNA-ДНК, образовавшийся
после выхода из этих кристаллических структур в белках P. Furiosus.
В составной структуре три области в форме полумесяца ДНК-лигазы P. Furiosus
окружают центральную дуплексную ДНК, окруженную замкнутым кольцом PCNA.
Взаимная ориентация доменов лигазы удивительно отличается от кристаллических структур
и, следовательно, большая перестройка домена происходит при участии тройного
комплекса формирования. В модели изображения с электронного микроскопа видны
контакты ДНК-лигаз и PCNA на двух областях, обычные PIP-боксы и новый, второй контакт в середине
аденилирования домена. Интересно также, что наблюдается значительный наклон ДНК
от оси кольца PCNA. На основе этого структурного анализа был установлен
механизм, в котором PCNA связывает и выпускает белок последовательно. В самом
деле, большинство белков, связываемых PCNA имеют одни и те же области
связывания в междоменной соединительной петле (IDCL) и общий С-концевой хвост
PCNA. Структурные особенности исключают возможность того, что три белка
связаются с одним кольцом PCNA одновременно, так как ДНК-лигаза занимает два из
трех субъединиц тримера PCNA.
В случае RFCPCNA-ДНК объединения, полученного таким же способом
электронного микроскопирования, RFC полностью закрывает кольцо PCNA, таким
образом блокируя доступ другим белкам. Эти тройные комплексы составлены в
пользу механизма, включающего последовательное связывание и высвобождение
факторов репликации.
12.
Флэп-эндонуклеаза 1 (FEN1)
Эффективная работа фрагментов Оказаки при создании непрерывной цепи ДНК
имеет важное значение для отстающих нитей синтеза в асимметричной репликации
ДНК. Праймазно синтезированные РНК / ДНК-праймеры должны быть удалены, чтобы
присоединиться к фрагментам Оказаки в интактную непрерывную нить ДНК.
Флэп-эндонуклеаза 1 (FEN1) в основном отвечает за эту задачу. Образование фрагментов
Оказаки сильно согласовано с непрерывным синтезом ДНК и взаимодействием
ДНК-полимеразы, FEN1 и ДНК-лигазы с PCNA, чтобы эти ферменты действовали
последовательно в ходе процесса образования, как описано выше. FEN1, структура
конкретной 5'-эндонуклеазы, в частности, признает двухцепочечную ДНК с
необозначенного 5' конца.
В эукариотических системах фрагменты Оказаки 5' конца ДНК образованы
активным замещением цепи ДНК-полимеразы δ. Lig I уплотняет окончание после схлопывания ДНК, расщеплённого
FEN1. Эти этапы обработки облегчаются при помощи PCNA. Взаимодействия между
эукариотическими FEN1 и PCNA были хорошо изучены, как и стимулирующее действие
PCNA на активность FEN1.
Кристаллическая структура человеческого FEN1-PCNA комплекса выявила три
молекулы FEN1, связанные с каждой субъединицей PCNA на триммерном кольце в
различных конфигурациях. В настоящее время на основе этого структурного анализа
вместе с описанием секции ДНК-лигазы изучается «флип-флоп» механизм перехода,
который позволяет белкам внутренне переключаться для выполнения различных
функций на этом же зажиме ДНК в настоящее время. Были найдены эукариотические
гомологи FEN1 у архей.
Были определены кристаллические структуры FEN1 из М. Jannaschii, P. Furiosus, P. Horikoshii, A. Filgidus, и С. Solfataricus. Кроме того, подробные биохимические
исследования проводились на FEN1 в P. Horikoshii. Таким образом, исследования белков
FEN1 у архей предоставили важные сведения о структурной основе реакции
расщепления и схлопывания ДНК. Наши последние исследования показали, что
активность флап-эндонуклеазы FEN1 в P. furiosus стимулировалась
PCNA.
Кроме того, в лабораторных условиях было доказано стимулирующее действие
PCNA на последовательные действия FEN1 и ДНК-лигазы. На основании этих
результатов была построена модель молекулярного механизма переключения из
последних частей фрагментов Оказаки. Четвертичный комплекс FEN1-Lig-PCNA-ДНК был выделен для
электронного микроскопирования частиц. Эти исследования позволят получить более
конкретный образ молекулярного механизма.
13.
Результаты и перспективы
Исследование молекулярного механизма репликации ДНК было центральной
темой молекулярной биологии. Археи стали популярными в общем секвенировании
генома, как описано выше, и большинство белков репликации ДНК в настоящее время
можно одинаково биохимическии характеризовать. Кроме того, исследования архей
особенно интересны для понимания механизмов, посредством которых клетки живут в
экстремальных условиях окружающей среды.
Кроме того, следует также отметить, что белки из гипертермофильных
архебактерий являются более стабильными, чем из мезофильных организмов, и они
являются предпочтительными для структурных и функциональных анализов
высокоорганизованных комплексов, таких как реплисомы. Для понимания каждого из
процессов, участвующих в метаболизме ДНК, необходимы исследования
высокоорганизованных комплексов, а не отдельных белков, и исследования архей
будут постоянно вносить вклад в развитие и продвижение полевых исследований
репликации ДНК, которые были обобщены в недавнем обзоре.
В дополнение к основным исследованиям в области молекулярной биологии,
репликации ДНК белков из термофилов были весьма полезными реагентами для генной
манипуляции, в том числе генетической диагностики, принудительного набора ДНК и
выявления бактериальных и вирусных инфекций, а также для фундаментальных
исследований. Многочисленные ферменты продавались во всем мире, и используются
по сегодняшний день. Примером успешной выработки ДНК-полимеразы архей для ПЦР
является создание слитого белка Pol B из P. furiosus и получение
неспецифического белка, связывающего двухцепочечную ДНК, Sso7d, из S.solfataricus,
при помощи методов генной инженерии.
Слитая ДНК-полимераза преодолела низкую процессивность дикого типа Pol B
высоким сродством Sso7d к ДНК. В качестве другого примера можно использовать
успешную разработку нового поступательного метода ПЦР с использованием архейных
Pol B с помощью мутантной PCNA. Были разработаны некоторые технологии
секвенирования ДНК, называемые «следующим поколением секвенирования», имеющиеся
в продаже в настоящее время.
Одномолекулярное обнаружение, применяющееся с помощью меченных красителем
модифицированных нуклеотидов, позволяющее читать большие длины, сейчас известно
как «третье поколение секвенирования ДНК». Эти технологии испльзуют
ДНК-полимеразы или ДНК-лигазы из различных источников, что говорит о том, что
эти ферменты репликации ДНК необходимы для развития технологии манипулирования
ДНК. Эти факты доказывают, что развитие фундаментальных исследований в области
молекулярной биологии репликации ДНК у архей будет способствовать развитию
новых технологий генной инженерии.
Linguistical Analysis of the Text
1. Terms
перевод специализированный текст английский
One of its most conspicuous features of the translated text
is the abundance of terms of different types in the sphere of genetics. While
translating them I had to refer to various translation techniques, some
examples of their types and the ways of their translation are introduced in the
table below.
Table 1.
|
Type of the term
|
Example
|
Translation
|
Translation
technique
|
|
Simple
|
ancestor
|
предок
|
Established
Equivalent
|
|
eukaryote
|
эукариоты
|
Transliteration
|
|
polymerase
|
полимераза
|
Naturalization
|
|
procaryote
|
прокариоты
|
Transliteration
|
|
genome
|
геном
|
Transliteration
|
|
Derived
|
duplication
|
дублирование
|
Established
Equivalent
|
|
transmission
|
передача
|
Established
Equivalent
|
|
replication
|
репликация
|
Naturalization
|
|
translation
|
трансляция
|
Naturalization
|
|
binding
|
связывание
|
Established
Equivalent
|
|
protein
|
белок
|
Established
Equivalent
|
|
site
|
сайт
|
Naturalization
|
|
|
|
|
|
Word Combination
|
protein factors
|
протеиновый фактор
|
Naturalization
|
|
domain of life
|
домен жизни
|
Calque
|
|
evolutional
perspective
|
эволюционная точка зрения
|
Calque
|
|
initiation
factor
|
фактор инициации
|
Semi-calque
|
|
Compound
|
homohexamer
|
гомогексамер
|
Transliteration
|
|
structure-function
|
структурно-функциональный
|
Naturalization
|
|
aminoacid
|
аминокислота
|
Semi-calque
|
2.
Abbreviations
An abbreviation (from Latin
<http://en.wikipedia.org/wiki/Latin> brevis, meaning short)
is a shortened form of a word or phrase
<http://en.wikipedia.org/wiki/Phrase>. Usually, but not always, it
consists of a letter <http://en.wikipedia.org/wiki/Letter_(alphabet)> or
group of letters taken from the word or phrase. For example, the word abbreviation
can itself be represented by the abbreviation abbr., abbrv. or abbrev.
Most of abbreviations that are used in the text
coincide with Russian biochemical terminology.
Table 2.
|
Abbreviation
|
Way of Word
Formation
|
Russian
Translation
|
Way of translation
|
|
DNA
|
Letter
abbreviation
|
ДНК (дезоксирибонуклеиновая
кислота)
|
The molecular
mechanism of DNA replication has been one of the central themes of
molecular biology
|
|
ORC
|
Letter
abbreviation
|
Точка инициации
транскрипции
|
…was completely
suppressed by binding to DNA containing the ORB.
|
|
RFC
|
Letter
abbreviation
|
Репликационный фактор С
|
Furthermore, the
direct interaction between PriSL and the clamp loader RFC
|
|
PriSL
|
Contraction and
Abbreviation
|
Праймаза SL
|
Furthermore, the
direct interaction between PriSL and the clamp loader RFC
|
|
GINS
|
Letter
abbreviation
|
Комплекс 5-1-2-3
|
…therefore, GINS
is an acronym for Japanese go-ichi-ni-san, meaning 5-1-2-3, after these four
subunits
|
|
AT-rich
|
Letter
abbreviation+suffix
|
Аденозин-тимин-богатые
|
… two of the
repeats are longer and surround apredicted DUE with an AT-rich
sequence
|
|
DUE
|
Letter
abbreviation
|
Элемент раскручивания ДНК
|
… two of the
repeats are longer and surround apredicted DUE with an AT-rich
sequence
|
|
ORB
|
Letter
abbreviation
|
Фактор инициации
распознавания
|
The longer
repeated sequence was designated as an ORB
|
|
RIP
|
Letter
abbreviation
|
Точка инициации репликации
|
…which was
determined earlier by an in vivo replication initiation point (RIP)
assay
|
|
ATP
|
Letter
abbreviation
|
АТФ (аденозинтрифосфорная
кислота)
|
…was not
drastically different in the presence and absence of ATP
|
|
OB
|
Letter
abbreviation
|
a structurally
similar domain containing a common fold, called the OB (oligonucleotide/oligosaccharide
binding)-fold
|
|
EM
|
Letter
abbreviation
|
Электронное
микроскопирование
|
…using the P.
furiosus proteins was obtained by EM single particle analysis.
|
3. Addition and Omission
the process of my translation I needed to employ various
translation techniques to transfer the meaning of the Source text in full
simultaneously keeping to the norms of Russian language. Some examples are
presented in the next part of the linguistic analysis.I had to add some words
in my Russian translation, and sometimes, vice versa I had to omit some words.
Some results are shown in the next two tables.
Table 3. Addition.
|
Example
|
Translation
|
|
In addition,
recent microbial ecology has revealed that archaeal organisms inhabit
not only extreme environments, but also more ordinary habitats
|
Кроме того, недавние
экологические исследования показали, что археи обитают не только в
экстремальных условиях, но и в более обычной среде обитания.
|
|
On the other
hand, it is also interesting that the circular genome structure is
conserved in Bacteria and Archaea and is different from the linear form of
eukaryotic genomes.
|
С другой стороны, интересным
является тот факт, что кольцевая структура генома сохраняется у бактерий
и архей и отличается от линейной формы эукариотических геномов.
|
|
Because Archaea
was recognized as the third domain of life later, the archaeal DNA
replication research became active after 1990.
|
Так как позже археи были
признаны третьим доменом жизни, исследования репликации ДНК архей стали активно
проводиться после 1990 года.
|
|
The interaction
between Cdc6/Orc1 and Mcm is probably general.
|
Взаимодействие между
Cdc6/Orc1 и MCM, вероятно, является общим для изучаемых
организмов.
|
|
These processing
steps are facilitated by PCNA.
|
Эти этапы обработки проходят
более легко при помощи PCNA.
|
Table 4. Omission.
|
ExampleTranslation
|
|
|
The accurate
duplication and transmission of genetic information are essential and
crucially important for living organisms.
|
Точное дублирование и
передача генетической информации являются абсолютно необходимыми для
живых организмов.
|
|
The archaeal
replication machinery is probably a simplified form of that in eukaryotes.
|
Механизм репликации у
архей, вероятно, более упрощен, чем у эукариот.
|
|
These two clamp
loaders adopt similar subunit organizations and spatial distributions, but
the functions of the individual subunits are likely to be diverse.
|
Эти два зажима принимают
аналогичные организации субъединиц и пространственное распределение, но функции
субъединиц отличаются.
|
4. Occasional contextual correspondence
Occasional contextual substitutions are used only for a given
occasion and not registered in dictionaries. That is why they are very
individual and depend on translator’s creative abilities.
5.
|
English sentence
|
Russian
translation
|
Dictionary
meaning
|
|
However, this
recruitment is not sufficient for the unwinding function of MCM, and some
other function remains to be identified for the functional loading of this
helicase to promote the progression of the DNA replication fork.
|
Однако, этого сигнала
недостаточно для активации функции раскручивания MCM, и некоторые другие
функции еще предстоит определить, чтобы функциональная нагрузка этой геликазы
содействовала прогрессированию вилки репликации ДНК.
|
recruitment [rɪ'kruːtmənt]
1) а) набор, вербовка, комплектование 2) восстановление здоровья, поправка 3)
подкрепление, усиление 4) увеличение численности популяции по мере взросления
потомства 5) пересадка тканей или клеток в пределах одного организма
|
|
However, this
idea is still not universal for the eukaryotic replicative helicase.
|
Однако эта идея все же не
распространяется на эукариотические репликативныхе геликазы.
|
universal [ˌjuːnɪ'vɜːs(ə)l]/
1) универсальный 2) глобальный, всемирный 3) всеобщий
|
|
Our
immunoprecipitation experiments showed that Mcm1 co-precipitated with Mcm3
and GINS, although they did not form a heterohexameric complex, suggesting
that Mcm1 is involved in the replisome or repairsome and shares some
function in T. kodakarensis cells.
|
Наши эксперименты показали,
что иммунопреципитация MCM1 сопоставима с MCM 3 и GINS,
и хотя они не образуют гетерогексамерного комплекса, предполагают, что MCM1
принимает участие в реплисоме или репарсоме и выполняет некоторые
функции в клетках Т. kodakarensis.
|
share разделять, делить;
совместно использовать (например, ресурс)
|
|
Although western
blot analysis could not detect Mcm2 in the extract from exponentially growing
T. kodakarensis cells, a RT-PCR experiment detected the transcript
of the mcm2 gene in the cells.
|
Хотя западный блот-анализ
не смог обнаружить MCM 2 в выписке из экспоненциально растущей клетке T.
kodakarensis, RT-PCR эксперимент обнаружил наличие следов
генетического кода MCM2 в клетках.
|
transcript 1) запись
(звучащей речи) 2) расшифровка (стенограммы)
|
5. Concretisation.
Sometimes while translating a text we need to refer to
concretization. Concretization is usage of a word with narrower meaning in TL,
in comparison with the one used in SL.
Table 6.
|
English sentence
|
Russian sentence
|
|
Domains I and II
adopt a fold found in the AAA+ family proteins.
|
Домены I и II принимают складчатую
структуру в семье AAA + белков.
|
|
Some other
protein factors may function in various archaea, for example a protein that
is distantly related to eukaryotic Cdt1, which plays a crucial role during MCM
loading in Eukaryota, exists in some archaeal organisms, although its
function has not been characterized yet.
|
Некоторые другие белковые
факторы могут действовать у различных архей, например белок, отдаленно
связаный с эукариотической Cdt1, который играет решающую роль во время комплектации
MCM у эукариот, существует у некоторых архей, хотя его функции еще не
были охарактеризованы.
|
6. Modulation
Modulation may be divided into: 1) cause-and-effect; 2)
metonymic; 3) paraphrase. According to Peter Newmark’s definition, modulation
occurs when the translator reproduces the message of the original text in the
TL text and TL may appear dissimilar in terms of perspective. There are just
few examples of paraphrase below.
Table 7.
|
Example
|
Translation
|
|
The DNA helicase
activity of the recombinant Mcm1 protein is strong in vitro,
and a distinct amount of the Mcm1 protein is present in T.
kodakarensis cells.
|
Активность ДНК геликазы
рекомбинантного белка MCM1 обладает высокой активностью в лабораторных
условиях, и различные
количества белка MCM1 присутствует в клетках T. kodakarensis.
|
|
Gins51 was
preferred over Gins15 because of the order of the name of GINS.
|
Было отдано предпочтение
белку GINS51, потому что GINS15 не
соответствовал порядку цифр в аббревиатуре GINS.
|
7. Interpretation.
The main function of interpretation is to make the Target
Text transparent. It is reached by refusing from the literal translation. My
attempt of this is illustrated in Table 8.
Table 8.
|
ExampleTranslation
|
|
|
A
characteristic of the oriC is the conserved 13 bp repeats, as predicted earlier by bioinformatics,
and two of the repeats are longer and surround apredicted DUE (DNA unwinding
element) with an AT-rich sequence in Pyrococcus genomes.
|
ORIC является сохраненными повторами 13 связывающих
белков, как предсказано ранее
биоинформатиками, и два из повторов больше и окружают предполагаемый DUE
(элемент раскручивания ДНК) с AT-богатых последовательностей в геномах Pyrococcus.
|
|
Mcm3 is relatively
abundant in the T. kodakarensis cells.
|
В клетках Т. Kodakarensis достаточно высокий уровень содержания МСМ3.
|
8. Part of speech replacement
Any translator faces the necessity of a entire rebuilding of
a sentence. Still, the cases where a sentence can be reconstructed by changing
the part of speech of just one word are very common. This method of the
translation is actualy called part of speech replacement. The replacement in
these cases helps to perfectly convey the meaning of the source text without
stylistic distortion. Some examples of the application of this technique are
presented in the Table below.
Table 9.
|
Example
|
Translation
|
Explanation
|
|
… in addition to
the eukaryotic primase, DNA polymerase, and DNA ligase; these are obviously
different from bacterial proteins and these proteins were biochemically
characterized.
|
… в дополнение к
эукариотической праймазе, ДНК-полимеразе и ДНК-лигазе; это явно отличает
их от бактериальных белков, и эти белки биохимически характеризуются.
|
Adjective is
translated by Verb
|
|
These features
have encouraged us to study archaeal DNA replication, in the hopes of gaining
fundamental insights into this molecular mechanism and its machinery from an evolutional
perspective.
|
Эти особенности побудили
нас изучить механизм репликации ДНК архей, в надежде получить фундаментальное
понимание этого молекулярного механизма и его технику с точки зрения
эволюции.
|
Adjective is
translated by Noun
|
|
The
structure-function relationships of the archaeal Mcms have been aggressively
studied using purified proteins and site-directed mutagenesis.
|
В настоящее время
проводится большое количество исследований структурно-функциональных
отношений в архейных MCM с использованием очищенных белков и
сайт-направленного мутагенеза.
|
Verb is
translated by noun
|
|
In contrast, the
Cdc6-2 protein stimulates the helicase activity of MCM in Thermoplasma
acidophilum.
|
В противоположность этому,
Cdc6-2 белок является стимулятором геликазной активности MCM в Thermoplasma
acidophilum.
|
Verb is
translated by Noun
|
|
Based on the recent comprehensive genomic
analyses, thirteen archaeal species have more than one mcm gene.
|
В результате последнего комплексного анализа генома установлено,
что тринадцать видов архей имеют более одного MCM-гена.
|
Participle II is
translated by Noun
|
|
However, many of
the mcm genes in the archaeal genomes seem to reside within mobile
elements, originating from viruses.
|
Тем не менее, многие из MCM
генов в геноме архей, кажется, находятся внутри подвижных элементов, имеющих
вирусное происхождение.
|
Noun is
translated by Adjective
|
9. Transposition of members of a sentence.
In the process of our work we had to employ various
transfomations, leading to the changes of the structure of the sentences. Some
examples of transposition of members of a sentence are given in the following
Table.
Table 10.
|
Example
|
Translation
|
|
A mechanism
was proposed in which ORC1 binds to all four ORBs to introduce a
higher-order assembly for unwinding of the DUE with alterations in both
topology and superhelicity.
|
Был предложен механизм, в котором ORC1 связывает все четыре ORB, чтобы
ввести более высокий порядок сборки для разматывания DUE, с
изменениями как в топологии, так и в суперспиральности.
|
|
Furthermore,
the crystal structures of S. solfataricus Cdc6-1 and Cdc6-3 (two
of the three Cdc6/Orc1 proteins in this organism) forming a heterodimer bound
to ori2 DNA (one of the three origins in this organism), and that of A.
pernix ORC1 bound to an origin sequence were determined.
|
Кроме того, установлено,
что кристаллическая структура Cdc6-1 и Cdc6-3 у С. solfataricus
(два из трех Cdc6/Orc1 белков в этом организме) с образованием гетеродимера
связана с ori2 ДНК (один из трех истоков в этом организме), а так же
была определена область ORC1 у А. Pernix, связанная с исходной
последовательностью.
|
10.
Non-finite forms of the verbs
The text contains large numbers of impersonal forms of the
verbs. Translating such constructions, I had to correctly identify their type
and function, otherwise it was very easy to make a significant mistake. Some
interesting cases are presented in the Table below.
Table 11.
|
Example
|
Translation
|
Function
|
|
The molecular
mechanism of DNA replication has been one of the central themes of molecular
biology, and continuous efforts to elucidate the precise molecular
mechanism of DNA replication have been made since the discovery of the double
helix DNA structure in 1953.
|
Молекулярный механизм
репликации ДНК был одной из центральных тем молекулярной биологии, поэтому
прилагались постоянные усилия для выяснения точного молекулярного
механизма репликации ДНК, которые проводились после открытия структуры двойной
спирали ДНК в 1953 году.
|
Infinitive
Indefinite Active in the function of the Attribute
|
|
Rapid progress
of whole genome sequence analyses has allowed us to perform
comparative genomic studies.
|
Быстрый прогресс и полный
анализ генома позволили нам выполнить сравнительные исследования
генома.
|
Infinitive
Indefinite Active in the function of the Complex Object
|
|
Archaeal cells
have a unicellular ultrastructure without a nucleus, resembling
bacterial cells …
|
Клетки архей имеют
одноклеточную ультраструктуру без ядра, напоминающую бактериальные
клетки …
|
Participle I in
the function of the Attribute
|
|
Therefore, the
archaeal organisms are good models to elucidate the functions of each
component of the eukaryotic type replication machinery complex.
|
Таким образом, организмы
архей могут служить хорошими примерами и разъяснить функции каждого из
компонентов эукариотического типа в комплексе механизмов репликации.
|
Infinitive
Indefinite Active in the function of the Attribute
|
|
They proposed
that an initiation factor recognizes the replicator, now referred to
as a replication origin, to start replication of the chromosomal DNA.
|
Они предположили, что
фактор инициации признает репликатор, который сейчас называют
инициатором репликации, чтобы начать репликацию хромосомной ДНК.
|
Participle II in
the function of the Attribute
|
|
An important step
in characterizing the initiation of DNA replication in Archaea is to
understand how the Cdc6/Orc1 protein recognizes the oriC region.
|
Важный шаг в
характеристике инициации репликации ДНК у архей - понять, как белок
Cdc6/Orc1 признает область ORIC.
|
Gerund in the
function of the Attribute
|
|
A specific site
in the oriC to start unwinding in vitro, was identified using
the protein prepared by a denaturation-renaturation procedure recently.
|
Определенный сайт в ORIC,
соответствующий раскручиванию в лабораторных условиях, был определен с
помощью белка, подготовленного к процедуре денатурации-ренатурации.
|
Participle I in
the function of the Adverbial Modifier of Manner
|
|
After
unwinding of the oriC
region, the replicative helicase needs to remain loaded to provide
continuous unwinding of double stranded DNA (dsDNA) as the replication forks
progress bidirectionally.
|
После разматывания области ORIC, репликативная геликаза должна
оставаться загруженной, чтобы обеспечить непрерывное раскручивание
двухцепочечной ДНК по мере продвижения вилки репликации в двух
направлениях.
|
Gerund in the
function of Adverbial Modifier of Time
|
|
The MCM protein
complex, consisting of six subunits (Mcm2, 3, 4, 5, 6, and 7), is known to
be the replicative helicase “core” in eukaryotic cells.
|
Комплекс белка MCM,
состоящий из шести субъединиц (Mcm2, 3, 4, 5, 6, 7), как известно -
«ядро» репликативной геликазы в эукариотических клетках.
|
Infinitive
Indefinite Active in the function of Complex Subject
|
|
In contrast to
the eukaryotic MCM, the archaeal MCMs, consist of a homohexamer or homo
double hexamer, having distinct DNA helicase activity by themselves in
vitro, and therefore, these MCMs on their own may function as the
replicative helicase in vivo.
|
В отличие от
эукариотических MCM, архейные MCM состоят из гомогексамера или двойного
гомогексамера, которые имеют различные активности геликазы ДНК сами по
себе в лабораторных условиях, и, следовательно, эти MCM сами по себе
могут функционировать как репликативная геликаза в естественных условиях.
|
Participle I in
the function of the Attribute
|
|
Functional
interactions between Cdc6/Orc1 and Mcm proteins need to be investigated
in greater detail to achieve a more comprehensive understanding of the
conservation and diversity of the initiation mechanism in archaeal DNA
replication.
|
Функциональное
взаимодействие между Cdc6/Orc1 и белками МСМ должно быть исследовано
более подробно для достижения более глубокого понимания сохранения и
разнообразия механизмов инициирования репликации ДНК у архей.
|
Indefinite
Infinitive Passive in the function of the Compound Verbal Modal Predicate
|
|
Phenotypic
analyses investigating the sensitivities of the Δmcm1 and Δmcm2 mutant
strains to DNA damage caused by various mutagens, as reported for other DNA
repair-related genes in T. kodakarensis, may provide a clue to
elucidate the functions of these Mcm proteins.
|
Фенотипический анализ
исследования чувствительности Δ MCM1 и Δ MCM2
мутантных штаммов на повреждения ДНК, вызванными различными мутагенами, как
сообщалось для других репараций ДНК генов, связанных в Т.kodakarensis,
может дать ключ к выяснению функции белков MCM.
|
Indefinite
Infinite Active in the function of the Attribute
|
Bibliography
“The mechanisms of DNA replication”, D.Stewart, 2013;
“Biochemical Pathways: An Atlas of Biochemistry and
Molecular Biology” Second Edition, G.Michal, D.Schomburg, 2012;
“Types of Transformation in Translation Process”,
M.V.Spasova, N.R.Lopatina, 2012;
“Tips For Graduates”, A.V. Bolshak, N.R.Lopatina, N.V.
Semerdjidi, 2012;
“New English-Russian Dictionary of Biology”,
O.Chibisova, 2009.://www.translate.google.ru/
Glossary
|
1. Accurate 2. Abundant 3.
Adenine 4. Adenosine triphosphate 5.
Adipocyte 6. Aerobe 7. Alcohol 8. Aldehyde 9.
Alginate 10. Alleles 11. Among 12. Ancestor 13.
Anomers 14. Antibody 15. Anticodon 16.
Antigen 17. Apoactivator
18. Assay 19. Assembly 20. Attenuator 21.
Autotroph 22. Auxin 23. Auxotroph 24.
Available 25. Base 26. Bilayer 27. Binding 28.
Biochemistry 29. Bioluminescence 30. Blastoderm 31.
Bore 32. Bound 33. Branchpoint 34.
Brevitoxin 35. Buffer 36. Calcite 37. Cancer 38. Carbohydrate
39. Carcinogen 40.
Carotenoid 41. Catabolism
42. Catalyst 43.
Caught up 44. Cept 45. Chelate 46. Chlorophyll 47.
Chloroplast 48. Chromatin
49. Chromatography 50.
Chromosome 51. Cistron 52. Coactivator 53.
Codon 54. Coenzyme 55. Cofactor 56.
Collagen 57. Comparative 58. Confirmed 59.
Confirming 60. Conformation
61. Conserved 62.
Contribute 63. Crystal 64. Cytochromes 65.
Cytosine 66. Dark reactions 67. Deamination 68.
Dehydrogenase 69. Denaturation
70. Described 71.
Designated 72. Determined
73. Dialysis 74.
Differentiation 75. Dimer 76. Dipole 77.
Distortion 78. Domain 79. Double helix 80.
Duplex 81. Efficiency 82. Eicosanoid 83.
Eluate 84. Elucidate
85. Enantiomorphs 86.
Encode 87. Encouraged 88. Endonuclease 89.
Enhancer 90. Entropy 91. Equilibrium 92.
Eukaryote 93. Exon 94. Exponentially 95.
Fermentation 96. Forks 97. Furanose 98.
Gaining 99. Gametes 100. Gel fitration chromatography
101. Gene 102.
Genome 103. Genotype
104. Glycogen 105.
Glycolysis 106. Glycoprotein
107. Golgi apparatus 108.
Guanine 109. Habitat 110. Hairpin loop 111.
Half-life 112. Helix 113. Heme 114. Hemoglobin 115.
Heterochromatin 116. Heteropolymer 117.
Heterotroph 118. Histones
119. Homologues 120.
Homozygous 121. Host cell
122. Hydrolysis 123.
Hydrophobic 124. Hyperthermophilic 125.
Identified 126. Iinhibition 127. Immunoglobulin 128.
In vitro 129. In vivo 130. Indicate 131.
Inducer 132. Induction 133. Inherited 134.
Interacts 135. Interferon
136. Introduce 137.
Intron 138. Investigating 139. Ion 140.
Isomerase 141. Ketone 142. Ketosis 143.
Kilobase 144. Kinase 145. Kinetochore 146.
Krebs cycle 147. Laminarin 148. Leader region 149.
Lectin 150. Ligand 151. Ligase 152. Linear 153.
Lipid 154. Lipopolysaccharide 155.
Luciferase 156. Lysosome
157. Machineries 158.
Marker 159. Meiosis 160. Membrane 161.
Merodiploid 162. Mesosome
163. Metabolism 164.
Metamorphosis 165. Metaphase
166. Microarray 167.
Mitosis 168. Mobile 169. Molecular weight 170.
Monolayer 171. Monomer 172. Multiple 173.
Mutagen 174. Mutagenesis
175. Mutant 176.
Mutarotation 177. Myosin 178. Nuclease 179.
Nucleohistone 180. Nucleoside
181. Nucleosome 182.
Nucleotide 183. Nucleus 184. Oligonucleotide 185.
Oligosaccharide 186. Oncogene 187.
Operon 188. Organelle
189. Origin 190.
Oxidation 191. Palindrome
192. Paralogs 193.
Participate 194. Pentose 195. Peptide 196.
Peptidoglycan 197. Phenotype 198.
Pheromone 199. Phosphodiester 200. Phospholipid 201.
Phosphorylation 202. Photosynthesis 203.
Phycocyanin 204. Physiological 205. Pigment 206.
Plaque 207. Plasmid 208. Polyamine 209.
Polymer 210. Polymerase
211. Polypeptide 212.
Polysaccharide 213. Porphyrin 214.
Predicted 215. Primer 216. Primosome 217.
Prokaryote 218. Promoter
219. Prophase 220.
Proposed 221. Proprotein
222. Prostaglandin 223.
Protamines 224. Protein 225. Proteoglycan 226.
Protist 227. Pseudocycle
228. Purine 229.
Puromycin 230. Pyranose
231. Pyrimidine 232.
Pyrophosphate 233. Quantitative 234. Rapid 235.
Recombinant 236. Recombination 237. Reconstitute 238.
Recruitment 239. Redox potential 240.
Referred 241. Relatively 242. Renaturation 243.
Repair synthesis 244. Repeats 245.
Replication fork 246. Replicative 247.
Replicon 248. Repressor
249. Required 250.
Revealed 251. Ribose 252. Semipermeable 253.
Sequence 254. Sigma factor
255. Signal sequence 256.
Signature 257. Silica 258. Similarities 259.
Similarity 260. Soluble protein 261. Somatic cell 262.
Splicing 263. Sporulation
264. Stereoisomers 265.
Steroids 266. Stimulated 267. Stimulation 268.
Substantial 269. Substrate
270. Subunit 271.
Synapse 272. Template
273. Ternary 274.
Terpenes 275. Tetramer
276. Thioester 277.
Thymidine 278. Thymine 279. Topoisomerase 280.
Transamination 281. Transcription 282.
Transformation 283. Transition state 284.
Translation 285. Triggers 286. Trypsin 287.
Tumorigenesis 288. Turnover
289. Unidentified 290.
Universal 291. Unwinding proteins 292.
Utilized 293. Viroids 294. Virus 295. Vitamin 296.
Wild-type gene 297. Winged 298.
Wobble 299. X-ray crystallography 300.
Zygote
|
Точный Обильный Аденин
Аденозинтрифосфат Адипоцит Аэроб Алкоголь Альдегид Альгинат Аллель Среди
Предок Аномер Антитело Антикодон Антиген Апоактиватор Анализ Сборка
Аттенюатор Автотроф Ауксин Ауксотроф Доступный База Двойной слой Обязательный
Биохимия Биолюминесценция Бластодерма Отверстие Связанный Ветвление
Бревитоксин Буфер Кальцит Рак Углевод Канцероген Каротиноид Катаболизм
Катализатор Догонять Принимать Хелат Хлорофилл Хлоропласт Хроматин
Хроматография Хромосома Цистрон Коактиватор Кодон Коэнзим Кофактор Коллаген
Сравнительный Подтвержденный Подтверждающий Структура Сохраненный
Способствовать Кристалл Цитохром Цитозин Темновая реакция Дезаминирование
Дегидрогеназа Денатурация Описано Назначенный Определенный Диализ
Дифференциация Димер Диполь Искажение Домен Двойной спирали Дуплекс
Эффективность Эйкозаноид Элюат Разъяснять Антиподы Кодировать Воодушевленные
Эндонуклеаза Усилитель Энтропия Равновесие Эукариоты Эксон Экспоненциально
Ферментация Вилка Фураноза Получение Половые клетки Гель-фильтрационная
хроматография Ген Геном Генотип Гликоген Гликолиз Гликопротеина Аппарат
Гольджи Гуанин Среда обитания Закрепляющаяся петля Период полураспада Спираль
Гем Гемоглобин Гетерохроматин Гетерополимеру Гетеротроф Гистоны Гомологи
Гомозиготный Клетка-хозяин Гидролиз Гидрофобный Гипертермофильными
Идентифицированный Ингибирование Иммуноглобулин В пробирке В естественных
условиях Указывать Индуктор Индукция Унаследованный Взаимодействует Интерферон
Вводить Интрон Расследование Ион Изомераза Кетон Кетоз Килобаза Киназа
Кинетохор Цикл Кребса Ламинарин Основной регион Лектин Лиганд Лигаза Линейный
Липид Липополисахарид Люцифераза Лизосома Механизм Маркер Мейоз Мембрана
Меродиплоид Мезосома Метаболизм Метаморфоза Метафаза Микроскопирование Митоз
Мобильный Молекулярный вес Монослой Мономер Множественный Мутаген Мутагенез
Мутант Мутаротации Миозин Нуклеаза Нуклеогистон Нуклеозид Нуклеосома
Нуклеотид Ядро Олигонуклеотидн Олигосахарид Онкоген Оперон Органелла
Происхождение Окисление Палиндром Паралоги Участвовать Пентоза Пептид
Пептидогликан Фенотип Феромон Фосфодиэфир Фосфолипид Фосфорилирование
Фотосинтез Фикоцианин Физиологический Пигмент Бляшка Плазмида Полиаминный
Полимер Полимераза Полипептид Полисахарид Порфирин Предсказанный Инициатор
Праймосома Прокариот Промоутер Профаза Предложенный Пропротеин Простагландин
Протамины Белок Протеогликан Простейшее Псевдоцикл Пурина Пуромицин Пираноза
Пиримидин Пирофосфат Количественный Быстрый Рекомбинант Рекомбинация
Восстанавливать Вербовка Окислительно-восстановительный потенциал Приглашение
Относительно Ренатурация Восстановительный синтез Повторы Репликационная
вилка Репликационный Репликон Репрессор Требуется Показали Рибоза
Полупроницаемый Последовательность Sigma-фактор Последовательность
сигналов Подпись Кремнезем Сходства Сходство Растворимый белок Соматическая
клетка Сращивание Спорообразование Стереоизомеры Стероиды Стимулированный
Стимуляция Существенный Подложка Подразделение Синапс Шаблон Тройной Терпен
Тетрамер Тиоэфир Тимидин Тимин Топоизомераза Трансаминирование Транскрипция
Трансформация Переходное состояние Перевод Триггеры Трипсин Онкогенез Оборот
Неопознанный Универсальный Амортизация белков Использованный Вироиды Вирус
Витамин Природный ген Крылатый Колебаться Рентгеновская кристаллография
Зигота
|